Caractéristiques et évolution des souches cliniques d’entérocoques résistantes à la vancomycine et/ou au linézolide isolées en France, 2006-2022
// Characteristics and evolution of vancomycin- and/or linezolid-resistant enterococci clinical isolates in France, 2006–2022
Résumé
Introduction –
De nombreuses épidémies impliquant des souches d’entérocoques résistantes à la vancomycine (ERV) ont été rapportées en France et dans le monde. De plus, l’émergence de souches d’entérocoques résistantes au linézolide (ERL) a récemment été décrite. L’objectif de ce travail est de décrire les principales caractéristiques des souches d’ERV et d’ERL isolées en France et reçues au Centre national de référence de la résistance aux antibiotiques entre 2006 et 2022.
Méthode –
Toutes les souches reçues ont été caractérisées phénotypiquement (MALDI-TOF, antibiogramme, concentration minimale inhibitrice – CMI) et génotypiquement (PCR en temps réel, séquençage complet du génome). Le typage et la comparaison des souches d’ERV ont été réalisés avec différentes techniques au cours du temps (électrophorèse en champ pulsé, rep-PCR, analyse génomique comparative ou spectroscopie infrarouge à transformée de Fourier).
Résultats –
Sur les 7 416 souches d’ERV reçues sur la période 2006-2022, une très grande majorité appartenait à l’espèce Enterococcus faecium (91,8-98,8%), suivie par E. faecalis (0,8-7,9%). Le gène vanA était majoritaire (76,5%), suivi du gène vanB (22,2%). La quasi-totalité des souches d’E. faecium (>95%) appartenaient au complexe clonal (CC) 17. Depuis 2017, il a été observé une augmentation significative des ERL liée à la diffusion dans certaines régions de souches épidémiques présentant une résistance chromosomique, mais aussi à l’acquisition de gènes plasmidiques (notamment optrA et poxtA) par des souches non clonales. Par typage moléculaire, il a été démontré que de nombreux clones d’ERV (majoritairement E. faecium) étaient sporadiques tandis que certains clones hyperépidémiques (isolés chez ≥5 patients) ont été détectés dans plusieurs établissements/villes différents avec une diffusion locorégionale.
Conclusion –
La majorité des souches d’ERV circulant en France sont des souches d’E. faecium adaptées à l’environnement hospitalier (CC17) et porteuses de l’opéron vanA. Il y a aussi une émergence alarmante des souches d’ERL qui doit être étroitement surveillée.
Abstract
Introduction –
Many hospital outbreaks involving vancomycin-resistant enterococci (VRE) have been reported in France and internationally. In addition, the emergence of linezolid-resistant enterococci (LRE) has recently been outlined. This study aimed to describe the main characteristics of VRE and LRE human isolates collected in France and received by the National Reference Centre for Antimicrobial Resistance between 2006 and 2022.
Method –
All strains received were characterized phenotypically (MALDI-TOF, antibiogram, MICs) and genotypically (real-time PCR, WGS). Typing and comparison between VRE isolates were performed using different techniques over time (pulsed-field gel electrophoresis, rep-PCR, comparative genomic analysis, Fourier transform infrared spectroscopy).
Results –
Out of 7,416 VRE clinical isolates received from 2006 to 2022, a large majority belonged to the species E. faecium (91.8–98.8%) followed by E. faecalis (0.8–7.9%). The vanA gene was in majority (76.5%) followed by vanB (22.2%). Almost all E. faecium isolates (>95%) belonged to the clonal complex (CC) 17. Since 2017, a significant increase of LRE has been noticed in association with the spread in some regions of epidemic strains exhibiting a chromosome-encoded resistance, but also with the acquisition of plasmid-mediated genes (particularly optrA and poxtA) by non-clonally-related strains. By molecular typing, it was pointed out that numerous VRE clones (mainly E. faecium) were sporadic whereas some hyperepidemic clones (isolated in ≥5 patients) were detected in several hospitals/cities with a loco-regional diffusion.
Conclusion –
The majority of human VRE isolates circulating in France are hospital-adapted E. faecium strains (CC17) harboring the vanA operon. There is also an alarming emergence of LRE isolates, which must be closely monitored.
Introduction
Bien que commensaux du microbiote intestinal, les entérocoques constituent une des principales causes d’infections associées aux soins (5-15%), et de nombreuses épidémies impliquant des souches d’entérocoques résistantes à la vancomycine (ERV) ont été rapportées 1,2. Aux États-Unis, près de 80% des isolats cliniques d’Enterococcus faecium sont résistants à la vancomycine tandis que la prévalence de ces souches en Europe varie considérablement d’un pays à l’autre, allant de moins de 1% (France) à plus de 50% en 2021 (Chypre, Malte ou Lituanie) 3,4. Chez les entérocoques, la résistance aux glycopeptides est due à l’acquisition d’opérons van codant pour des enzymes permettant la synthèse de précurseurs du peptidoglycane de faible affinité pour ces antibiotiques qui préviennent la formation des précurseurs naturels 5. À l’échelle mondiale, les opérons vanA et vanB sont de loin les plus fréquents parmi les souches cliniques d’ERV 6. Pour rappel, vanA confère classiquement une résistance de haut niveau à la vancomycine et à la téicoplanine (concentration minimale inhibitrice – CMI >32 mg/L) tandis que vanB est responsable d’une résistance variable à la vancomycine (CMI de 4 à >32 mg/L) avec sensibilité conservée à la téicoplanine (CMI <2 mg/L) 5,6. Le succès épidémiologique des souches d’ERV chez E. faecium est principalement dû à la dissémination internationale d’un complexe clonal particulier, appelé CC17, qui comprend des souches cliniques caractérisées par une résistance de haut niveau aux pénicillines et aux fluoroquinolones, la détection fréquente de gènes de virulence (esp, hyl) et la présence d’une séquence d’insertion spécifique (IS16) 7,8. Plus récemment, il a été proposé que ce CC17 fasse partie d’une lignée génétique (clade A1) comprenant des souches humaines hospitalières multirésistantes aux antibiotiques ayant émergé d’une autre lignée génétique associée aux animaux (clade A2) après l’introduction des antibiotiques en médecine et l’utilisation de dérivés glycopeptidiques (avoparcine) comme facteurs de croissance en élevage. Cette autre lignée diffère des souches humaines commensales (clade B) 9. Depuis quelques années, l’isolement et l’identification des souches d’entérocoques résistantes au linézolide (ERL) sont de plus en plus rapportés, notamment en relation avec la diffusion de gènes transférables (cfr-like, optrA et poxtA) portés par des plasmides 10,11.
Cet article décrit les caractéristiques phénotypiques et génotypiques des souches d’ERV et d’ERL isolées en France et reçues au Centre national de référence (CNR) de la résistance aux antibiotiques, entre 2006 et 2022.
Matériel-Méthodes
Les souches d’entérocoques ont été reçues au CNR entre 2006 et 2022. L’envoi de ces souches se fait sur la base du volontariat dans le cadre d’une demande spécifique (caractérisation de résistance et/ou comparaison de souches), il n’y a aucune obligation. Elles ont toutes été caractérisées phénotypiquement et génotypiquement. L’identification au niveau de l’espèce bactérienne a été réalisée par spectrométrie de masse MALDI-TOF (Microflex, Bruker Daltonics) et par séquençage des gènes sodA ou rrs (ARNr 16S) si nécessaire. La sensibilité in vitro aux antibiotiques (ampicilline, gentamicine, érythromycine, clindamycine, quinupristine-dalfopristine, vancomycine, téicoplanine, norfloxacine, lévofloxacine, linézolide, cotrimoxazole et chloramphénicol) a été évaluée par la méthode des disques sur milieu gélosé selon les recommandations du Comité de l’antibiogramme de la Société française de microbiologie et du European Committee on Antimicrobial Susceptibility Testing. Depuis 2017, les CMI de différents antibiotiques (vancomycine, téicoplanine, daptomycine, tigécycline, linézolide, tédizolide, dalbavancine, télavancine et oritavancine) sont déterminées par la méthode de microdilution en milieu liquide (Sensititre, ThermoFisher).
La détection des gènes de résistance aux glycopeptides les plus fréquents (vanA, vanB, vanC1, vanC2/C3 et vanD) a été réalisée par PCR en temps réel multiplex. En cas de résultat négatif, les autres gènes de l’alphabet van (vanE, vanG, vanL, vanM et vanN), plus rares, sont recherchés secondairement par PCR en temps réel monoplex. La détection des gènes de résistance au linézolide (cfr-like, optrA et poxtA) a également été réalisée par PCR en temps réel. Le gène ptsD, potentiellement impliqué dans le pouvoir de diffusion des souches d’E. faecium, ainsi que la séquence d’insertion IS16, spécifique du CC17, ont été recherchés par PCR en temps réel.
Le typage et la comparaison des souches d’ERV ont été réalisés avec différentes techniques selon la période. Pendant des années, la grande majorité des investigations a été réalisée par électrophorèse en champ pulsé (ECP ou PFGE en anglais pour Pulsed-Field Gel Electrophoresis) après macrorestriction de l’ADN total par l’endonucléase SmaI (2006-2012 et 2017-2019), progressivement remplacée par d’autres méthodes génomiques : rep-PCR (Diversilab, bioMérieux) (2012-2016) et analyse génomique comparative par séquençage entier du génome (WGS pour Whole Genome Sequencing) (depuis 2019). Les données WGS permettent de déterminer le Sequence Type (ST) par approche MLST in silico, et surtout d’effectuer une analyse phylogénétique par analyse des SNP (Single Nucleotide Polymorphism) sur core genome (cgSNP). Depuis 2020, l’utilisation de l’instrument IR biotyper (Bruker), basée sur la technologie de de la spectroscopie infrarouge à transformée de Fourier (IRTF), a aussi permis d’effectuer des comparaisons de souches dans le cadre d’épidémies. Le séquençage entier du génome a aussi été utilisé pour étudier les gènes et les mutations de résistance aux antibiotiques, ainsi que leurs support et environnement génétiques.
Résultats
Après un pic en 2008, le nombre de souches d’ERV reçues (sur la base du volontariat) et analysées au CNR s’était stabilisé entre 2009 et 2014 (avec une moyenne de 300 à 350 souches par an) avant de fortement augmenter entre 2015 et 2019 (x 3,1) (figure 1a). Sur les 7 416 souches d’ERV reçues pour expertise sur la période 2006-2022, une très grande majorité appartenait à l’espèce E. faecium (de 91,8 à 98,8%), suivie par E. faecalis (de 0,8 à 7,9%). Les autres espèces (E. avium, E. casseliflavus, E. durans, E. gallinarum, E. gilvus, E. hirae et E. raffinosus) étaient faiblement représentées (figure 1b, tableau). Ces souches ont principalement été isolées d’écouvillonnages anorectaux ou de selles lors de dépistages (>87%) tandis que peu de souches responsables de bactériémies ou d’infections urinaires ont été reçues (respectivement 1,3 et 2,3%). Les souches d’ERV ont été isolées dans de nombreux types de services cliniques, dont les principaux (5 à 15% selon les années) sont les services de réanimation, de néphrologie-dialyse, d’hépato-gastro-entérologie et de médecine interne/maladies infectieuses.
 Agrandir l'image
Agrandir l'image Agrandir l'image
Agrandir l'imageConcernant les mécanismes de résistance, le gène vanA était toujours majoritaire parmi les souches d’ERV sur la période 2006-2022 (76,5%), suivi du gène vanB (22,8%) et du gène vanD (n=67, 0,9%) (figure 1c, tableau). Quelques souches étaient porteuses des deux gènes vanA et vanB (n=30, 0,4%). Les gènes vanG et vanN ont été identifiés une seule fois chacun et la présence du gène vanM a été notée chez deux souches d’E. faecium (tableau).
Par détection de l’IS16, une grande majorité (>95%) des souches d’E. faecium ont pu être identifiées comme appartenant au CC17. De la même façon, le gène ptsD, potentiellement impliqué dans le pouvoir épidémique des souches hospitalières d’E. faecium, a été retrouvé chez de nombreuses souches (>90%).
Toutes les souches d’E. faecalis étaient sensibles à l’ampicilline alors qu’environ un tiers d’entre elles (entre 22 et 42% selon les années) étaient catégorisées résistantes à haut niveau à la gentamicine. Parmi les souches d’E. faecium résistantes aux glycopeptides testés, la grande majorité (>96%) étaient résistantes à l’ampicilline alors que la résistance de haut niveau à la gentamicine était présente chez 48 à 69% des souches selon les années, sans qu’une tendance au cours du temps ne soit observée. Chez ces deux espèces, cette résistance était due à la production de l’enzyme bi-fonctionnelle AAC(6’)-APH(2’’) dont le gène a été retrouvé par PCR ou WGS.
Alors que le phénomène était peu retrouvé auparavant, le nombre de souches d’ERL (CMI allant de 8 à >256 mg/L) a nettement augmenté à partir de 2017 (figure 1d). Les deux espèces concernées étaient surtout E. faecium et E. faecalis. Parmi les ERL, la résistance des souches d’E. faecalis était principalement d’origine plasmidique (notamment optrA). Au contraire, chez E. faecium, des mutations chromosomiques dans le gène de l’ARNr 23S étaient le mécanisme de résistance principal.
Les souches d’ERL chez E. faecalis sont quasi-exclusivement sensibles aux glycopeptides, alors que celles appartenant à l’espèce E. faecium sont principalement des ERV de génotype vanA. Seules quelques souches (n=33) résistantes à la tigécycline (CMI de 0,5 à 3 mg/L) ont été identifiées, alors que la résistance de haut niveau à la daptomycine (CMI >8 mg/L) reste exceptionnelle en France (n=30, soit 0,3%), ces souches étant plus généralement des ERV. Enfin quelques souches d’E. faecium (vanA ou vanB) étaient corésistantes à l’ampicilline, à la vancomycine (CMI de 32 à >256 mg/L), à la daptomycine (CMI=8 mg/L) et au linézolide (CMI=8-32 mg/L).
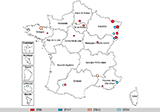
Par typage moléculaire, il a été démontré que de nombreux clones d’ERV (très majoritairement appartenant à l’espèce E. faecium) étaient sporadiques tandis que dans les établissements où plusieurs clones étaient rapportés, il existait généralement un clone majoritaire et plusieurs clones minoritaires. Des clones (vanA ou vanB) pouvant être considérés comme hyperépidémiques (isolés chez ≥5 patients) ont été détectés dans plusieurs établissements/villes différents au sein d’une même région. Sur la période 2015-2022, les principales régions touchées par des épidémies à E. faecium vanA étaient l’Île-de-France, les Hauts-de-France, le Grand Est et la Bretagne. Concernant les épidémies à E. faecium vanB, les régions du Grand Est, de la Nouvelle-Aquitaine et de la Provence-Alpes-Côte d’Azur étaient les plus atteintes. Sur 153 souches d’E. faecalis dont le génome était séquencé, les principaux clones appartenaient aux ST suivants : ST16 (18%), ST40 (18%), ST179 (13%) et ST480 (8%). Chez les 848 souches d’E. faecium séquencées, une diffusion majoritaire du ST80 a été notée à l’échelle du territoire national (37%), suivi des ST117 (16%) et ST612 (10%) (figure 2). D’autres ST moins fréquents ont été identifiés, comme le ST78 (4%), le ST1478 (3%), le ST203 (3%), le ST761 (3%) et le ST18 (2%).
 Agrandir l'image
Agrandir l'imageDiscussion
Détectés de façon ubiquitaire dans la nature, les entérocoques sont généralement considérés comme des espèces commensales 12. Cependant, ils sont devenus des pathogènes humains opportunistes majeurs, avec en majorité l’implication des espèces E. faecalis et E. faecium dans les infections acquises à l’hôpital 2. En France, les souches d’E. faecium résistantes à la vancomycine sont classées comme bactéries hautement résistantes aux antibiotiques émergentes (BHRe), ce qui impose des stratégies de prévention et de contrôles spécifiques 13. Les entérocoques sont capables de survivre et de croître dans des environnements hostiles comme le tube digestif d’un patient hospitalisé sous traitement antibiotique 14. Ceci est dû à leur résistance naturelle à différentes classes d’antibiotiques (céphalosporines, aminosides à bas niveau, sulfamides) et à une remarquable plasticité génomique leur permettant l’acquisition de nombreux éléments génétiques mobiles transférables 15. C’est notamment le cas pour E. faecium qui peut acquérir de nombreux gènes de résistances (ex. vanA, vanB) portés par des transposons généralement plasmidiques 6. Depuis le début des années 90, il y a donc la dissémination de souches hospitalières d’E. faecium (CC17 ou clade A1) multirésistantes aux antibiotiques. Ceci explique la large prédominance de cette espèce parmi les souches cliniques d’ERV circulant en France et à l’étranger, comme en témoignent les nombreuses études épidémiologiques 16.
Ici, l’opéron de résistance vanA est majoritaire parmi les souches d’ERV, ce qui est en accord avec les études épidémiologiques conduites dans d’autres pays européens 6,15. Toutefois, dans certains pays (ex : l’Australie), les souches vanB sont majoritaires. Les souches d’entérocoques appelées VVE (pour Vancomycin-Variable Enterococci) sont des souches sensibles phénotypiquement aux glycopeptides alors qu’elles portent un opéron van qui n’est pas exprimé du fait de l’absence du système à deux composants vanRS, lequel régule l’expression de l’opéron de résistance. Elles ont déjà été décrites d’abord au Canada puis dans quelques autres pays (Australie, Corée du Sud, Danemark, Norvège) 17, et quelques-unes (n=10) ont été identifiées (mais pas encore publiées) au CNR en France. La présence de ces souches doit être signalée car elles sont capables de réverter sous pression de sélection antibiotique en redevenant résistantes à la vancomycine 18.
Alors qu’aucune souche d’E. faecalis résistante à l’ampicilline n’a été décrite jusqu’à ce jour en France et en Europe, la quasi-totalité des souches d’E. faecium résistantes à la vancomycine le sont à haut niveau. Ceci est en accord avec le fait que la grande majorité de ces souches appartiennent au CC17. En effet, les souches CC17 présentent des caractéristiques communes, dont la résistance à haut niveau aux pénicillines et aux fluoroquinolones 6,12,16. Environ un tiers des souches d’E. faecalis et environ deux tiers des souches d’E. faecium ont été catégorisées résistantes à haut niveau à la gentamicine, ce qui est similaire à ce qui est rapporté au niveau européen dans EARS-Net (1). Cette résistance est bien connue et est due à la production de l’enzyme bi-fonctionnelle AAC(6’)-APH(2’’) 6.
Depuis quelques années, les souches d’ERL sont de plus en plus rapportées. Cette émergence, qui constitue une situation préoccupante, est en partie liée à la diffusion de gènes transférables portés par des plasmides (notamment optrA et poxtA). Le gène optrA est d’ailleurs principalement détecté chez E. faecalis (généralement sensible aux glycopeptides) et poxtA l’est chez E. faecium (majoritairement vanA) 19,20. Cette diffusion récente des ERL en France pourrait être liée à l’utilisation de plus en plus importante du linézolide en médecine humaine (du fait de la mise sur le marché récente de formes génériques moins coûteuses) et d’une cosélection avec les phénicolés utilisés (florfénicol) en milieu vétérinaire (du fait d’une résistance croisée induite par ces gènes vis-à-vis de ces deux classes antibiotiques) et d’épisodes de transmission croisée en milieu hospitalier qui amplifient le phénomène 10,11,18. Le principal risque est de voir circuler des souches d’E. faecium résistantes à l’ampicilline, à la vancomycine et au linézolide ainsi que des plasmides co-portant des gènes de résistance à ces deux antibiotiques.
La résistance à la tigécycline, due à des mutations chromosomiques dans le gène rpsJ codant pour la protéine ribosomale S10 21 est rare. Enfin, l’isolement de souches résistantes à haut niveau à la daptomycine reste exceptionnel en France. La résistance à ces deux antibiotiques est également rarement observée dans les autres pays européens 10, mais elle doit continuer à être surveillée. L’approche WGS permet l’étude approfondie des souches présentant des phénotypes de résistance rares ou émergents, comme l’étude des mécanismes de résistance au linézolide, à la tigécycline ou à la daptomycine.
L’étude épidémiologique des ERV en France a permis de mettre en évidence de nombreuses épidémies d’ampleur plus ou moins importante au sein d’un ou de plusieurs établissements (souvent dans les mêmes régions), soulignant ainsi une diffusion locorégionale déjà précédemment décrite 22. Par ailleurs, certains de ces clones peuvent être retrouvés pendant plusieurs années, sans que le réservoir environnemental n’ait été identifié. Tel était le cas pour l’épidémie que le Grand Est a connue entre 2017 et 2019. Une situation similaire a été décrite en Suisse avec une épidémie à ERV de type vanB ayant duré de 2017 à 2020 23. Une grande majorité des différents ST détectés en France sont aussi retrouvés au niveau international, soulignant la large dissémination de souches d’entérocoques hospitalières et multirésistantes aux antibiotiques 24,25. La principale limite de cette étude est que l’envoi des souches au CNR s’effectue sur la base du volontariat et qu’il y a de ce fait un biais de recrutement.
Conclusion
La très grande majorité des souches d’ERV circulant en France sont des souches d’E. faecium adaptées à l’environnement hospitalier appartenant au clade A1 (anciennement CC17) et multirésistantes aux antibiotiques. Ces souches sont pour la plupart porteuses de l’opéron vanA. Ces ERV sont responsables d’épidémies dans de nombreux hôpitaux, avec des régions plus touchées que d’autres. Des clones considérés comme « hyperépidémiques » ont aussi été détectés dans plusieurs établissements/villes différents au sein d’une même région. En parallèle des ERV, il y a aussi l’émergence des souches d’ERL, qui portent des gènes de résistance transférables. Ce phénomène doit être étroitement surveillé, à la fois en médecine humaine et à la fois en médecine vétérinaire, du fait de la cosélection possible par des antibiotiques utilisés dans ces deux secteurs. Dans ce contexte, le rôle du CNR de la résistance aux antibiotiques (laboratoire associé Entérocoques) est majeur, et il est recommandé d’y adresser toute souche suspecte d’être un ERL, ainsi que toutes les souches avec des profils atypiques ou exceptionnels de résistance aux antibiotiques (comme les souches d’E. faecalis suspectes d’être résistantes à l’ampicilline, les souches résistantes à la tigécycline ou à la daptomycine, les souches VVE).
Remerciements
Nous remercions chaleureusement les nombreux collègues qui ont adressé leurs souches au CNR et ont ainsi permis de dresser un tableau de la situation des ERV et des ERL en France.
Liens d’intérêt
Les auteurs déclarent ne pas avoir de liens d’intérêt au regard du contenu de l’article.