Chronique
d'un début
de pandémie
Coordination scientifique : Jean-Claude Desenclos et Hélène Therre, Institut de veille sanitaire, Saint-Maurice, France.
- ÉDITORIAL 1/9 Grippe A(H1N1)v : L'Institut de veille sanitaire face à la pandémie The French Institute for Public Health Surveillance faces the Influenza A(H1N1)v pandemic
- PERSPECTIVE 2/9 Que nous apprennent les pandémies du passé ? What do we learn from pandemics of the past ?
- ARTICLES ORIGINAUX 3/9 Le point sur le virus de la nouvelle grippe A(H1N1)v Update on the new A(H1N1)v influenza virus
- 4/9 Cas d'infection par le nouveau virus de la grippe A(H1N1)v en France, situation au 5 juin 2009 Cases of infection with the new influenza A (H1N1)v virus in France, situation as of 5 June 2009
- 5/9 VoozaFlu : un outil au service de la surveillance de la nouvelle grippe A(H1N1)v VoozaFlu: a tool for monitoring the new influenza A(H1N1)v
- 6/9 Le point sur les paramètres épidémiologiques dans l’épidémie due au nouveau virus de la grippe A(H1N1)v Update on the epidemiological parameters in the epidemics caused by the new influenza A(H1N1)v virus.
- 7/9 Adaptation du dispositif de surveillance à la situation épidémiologique Adjusting the surveillance system to the epidemiological situation
-
8/9
Manifestations cliniques observées dans les premières séries de cas de grippe due au virus émergent Influenza
A(H1N1)v Clinical manifestations observed in the first groups of patients affected by the new influenza A(H1N1)v virus - 9/9 Grippe A(H1N1)v : naissance d’une pandémie - Bilan mondial au 5 juin 2009 Influenza A(H1N1)v: Birth of a pandemic – Global review as of 5 June 2009
 Le point sur le virus de la nouvelle grippe A(H1N1)v
Le point sur le virus de la nouvelle grippe A(H1N1)v
Introduction
Les virus influenza A sont classés en sous-types sur la base de leurs deux glycoprotéines d’enveloppe, l’hémagglutinine (HA) et la neuraminidase (NA). Chez les oiseaux aquatiques, qui constituent le réservoir de la diversité génétique des virus influenza A, 16 espèces d’hémagglutinine (H1 à H16) et neuf espèces de neuraminidase (N1 à N9) ont été répertoriées à ce jour. Chez différentes espèces de mammifères domestiques comme le porc ou le cheval, certains sous-types de virus influenza A se sont établis, donnant lieu à une transmission au sein de l’espèce et à des foyers d’infection et des épizooties. L’adaptation à l’espèce humaine de nouveaux sous-types viraux est à l’origine des pandémies de grippe. Ainsi, des virus de sous-type H1N1, H2N2 et H3N2 ont été introduits dans la population humaine respectivement en 1918 (grippe espagnole), 1957 (grippe asiatique) et 1968 (grippe de Hong Kong). Depuis la réintroduction, lors de la grande épidémie de grippe Russe russe, de virus H1N1 qui circulaient chez l’homme dans les années 1950, deux sous-types de virus grippaux A(H3N2) et A(H1N1) sont responsables des épidémies de grippe saisonnière.
Virus grippal chez le porc
Chez le porc, les premiers virus isolés en 1930 par Robert Shope sont de sous-type H1N1 et constituent le lignage dit « porcin classique » [1]. Ces virus ont vraisemblablement été introduits chez le porc à partir de l’homme lors de la pandémie de 1918. D’autres virus d’origine humaine ou aviaire, notamment de sous-type H3N2 et H1N1, ont été introduits chez le porc, établissant des lignages de virus porcins dits d’origine «humaine» ou «aviaire», respectivement. Du fait d’une évolution indépendante des virus porcins selon la localisation géographique, on distingue également des lignages nord- américain ou eurasiatique. La co-circulation de virus de différentes origines chez le porc a donné lieu à des événements de réassortiment par échange de segments génomiques dérivés de différents lignages. Ainsi, différents virus de sous-type H1N1, H1N2 et H3N2 co-circulent aujourd’hui chez le porc tant sur le continent nord-américain qu’en Europe et en Asie [2-4].
Transmission de virus porcins à l’homme
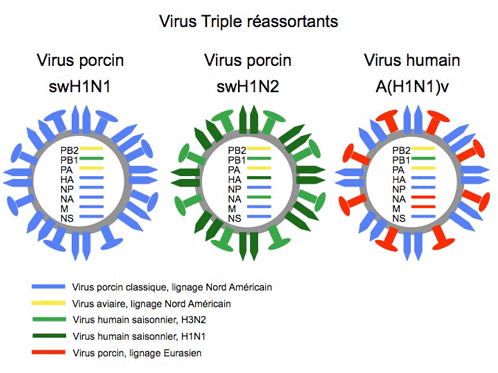
Des cas de transmission de virus porcins à l’homme, pour la plupart de sous-type H1N1 et plus rarement de sous-type H3N2, ont été rapportés à de multiples reprises [5]. Tous avaient pour origine un contact avec des porcs, et des analyses sérologiques ont mis en évidence un risque accru chez les professionnels en contact avec les porcs [6,7]. Ces cas sporadiques, généralement bénins, n’ont pas donné lieu à l’établissement d’une transmission interhumaine. La seule exception concerne l’épisode survenu en 1976 à Fort Dix, New Jersey, États-Unis, au cours duquel une transmission interhumaine significative d’un virus porcin H1N1 a donné lieu à 230 cas d’infection dont 12 hospitalisations et un décès, bien qu’aucun contact avec des porcs n’ait été documenté [8]. Aux États-Unis, entre décembre 2005 et février 2009, 11 cas humains sporadiques d’infection respiratoire par un virus porcin ont été détectés dont un sans lien établi avec des porcs [9]. Les virus en cause, dits « triple réassortants » prédominants dans la population porcine aux États-Unis, possèdent des segments dérivés du lignage porcin classique nord-américain (segments NP, M, NS), du lignage aviaire nord-américain (segments PB2, PA) et du lignage humain H3N2 (segment PB1), associés aux segments H1 et N1 du lignage porcin classique pour les virus H1N1 (10 cas) ou aux segments H1 et N2 des lignages humains H1N1 et H3N2, respectivement pour les virus H1N2 (1 cas).
Figure : Origine des segments génomiques des virus porcins à l’origine de cas d’infection chez l’homme aux États-Unis et du nouveau virus A(H1N1)v
Origine porcine des nouveaux virus A(H1N1)v
Des nouveaux virus A(H1N1)v d’origine porcine, identifiés initialement aux États-Unis chez deux patients de retour du Mexique, se sont avérés également responsables d’une épidémie atypique de grippe au Mexique. Dotés d’une capacité de transmission interhumaine avérée, ils ont diffusé au Canada, en Europe, en Amérique latine, au Japon et en Australie et sont aujourd’hui détectés dans 69 pays. L’analyse génétique des nouveaux virus A(H1N1)v a révélé qu’ils partagent les segments des virus porcins H1N1 « triple réassortants », mais qu’ils dérivent leurs segments NA et M de virus porcins appartenant au lignage eurasiatique (figure). Cette combinaison inédite de segments n’a jamais été identifiée auparavant chez les virus porcins ou humains.
![]()
- 1
- 2
- Vincent Enouf 1,
- Maude Bouscambert-Duchamp 2,
- Martine Valette 2,
- Ana Burgière 3,
- Valérie Caro 4,
- Jean-Claude
Manuguerra 3, - Bruno Lina 2,
- Sylvie van der Werf 1
1/ Centre national de référence du virus influenzae (Région-Nord), Institut Pasteur, Paris, France
2/ Centre national de référence du virus influenzae (Région-Sud), CHU de Lyon, Lyon, France
3/ Cellule d'intervention biologique d'urgence, Institut Pasteur, Paris, France
4/ Plateforme de génotypage des pathogènes et santé publique (PF8), Institut Pasteur, Paris, France
RÉFÉRENCES
 (1) Shope RE. The Etiology of Swine Influenza. Science 1931;73(1886) 214-5.
(1) Shope RE. The Etiology of Swine Influenza. Science 1931;73(1886) 214-5.
 (2) Vincent AL, Ma W, Lager KM, Janke BH, Richt JA. Swine influenza viruses a North American perspective. Adv Virus Res. 2008;72:127-54.
(2) Vincent AL, Ma W, Lager KM, Janke BH, Richt JA. Swine influenza viruses a North American perspective. Adv Virus Res. 2008;72:127-54.
 (3) Brown IH. The epidemiology and evolution of influenza viruses in pigs. Vet Microbiol. 2000;74(1-2):29-46.
(3) Brown IH. The epidemiology and evolution of influenza viruses in pigs. Vet Microbiol. 2000;74(1-2):29-46.
 (4)Van Reeth K. Avian and swine influenza viruses: our current understanding of the zoonotic risk. Vet Res. 2007;38(2):243-60.
(4)Van Reeth K. Avian and swine influenza viruses: our current understanding of the zoonotic risk. Vet Res. 2007;38(2):243-60.
 (5) Myers KP, Olsen CW, Gray GC. Cases of swine influenza in humans: a review of the literature. Clin Infect Dis. 2007;44(8):1084-8.
(5) Myers KP, Olsen CW, Gray GC. Cases of swine influenza in humans: a review of the literature. Clin Infect Dis. 2007;44(8):1084-8.
 (6) Olsen CW, Brammer L, Easterday BC, Arden N, Belay E, Baker I, Cox NJ. Serologic evidence of H1 swine Influenza virus infection in swine farm residents and employees. Emerg Infect Dis. 2002;8(8):814-9
(6) Olsen CW, Brammer L, Easterday BC, Arden N, Belay E, Baker I, Cox NJ. Serologic evidence of H1 swine Influenza virus infection in swine farm residents and employees. Emerg Infect Dis. 2002;8(8):814-9
 (7) Ayora-Talavera G, Cadavieco-Burgos JM, Canul-Armas AB. Serologic evidence of human and swine influenza in Mayan persons. Emerg Infect Dis. 2005;11(1):158-61.
(7) Ayora-Talavera G, Cadavieco-Burgos JM, Canul-Armas AB. Serologic evidence of human and swine influenza in Mayan persons. Emerg Infect Dis. 2005;11(1):158-61.
 (8) Gaydos JC, Hodder RA, Top FH Jr, Soden VJ, Allen RG, Bartley JD, et al. Swine influenza A at Fort Dix, New Jersey (January-February 1976). I. Case finding and clinical study of cases. J Infect Dis. 1977;136 Suppl:S356-S362.
(8) Gaydos JC, Hodder RA, Top FH Jr, Soden VJ, Allen RG, Bartley JD, et al. Swine influenza A at Fort Dix, New Jersey (January-February 1976). I. Case finding and clinical study of cases. J Infect Dis. 1977;136 Suppl:S356-S362.
 (9) Shinde V, Bridges CB, Uyeki TM, Shu B, Balish A, Xu X, et al. Triple-Reassortant Swine Influenza A (H1) in Humans in the United States, 2005-2009. N Engl J Med. 2009 May 22. [Epub ahead of print].
(9) Shinde V, Bridges CB, Uyeki TM, Shu B, Balish A, Xu X, et al. Triple-Reassortant Swine Influenza A (H1) in Humans in the United States, 2005-2009. N Engl J Med. 2009 May 22. [Epub ahead of print].
 (10) Novel Swine-Origin Influenza A (H1N1) Virus Investigation Team. Emergence of a Novel Swine-Origin Influenza A (H1N1) Virus in Humans. N Engl J Med. 2009 June 3. [Epub ahead of print].
(10) Novel Swine-Origin Influenza A (H1N1) Virus Investigation Team. Emergence of a Novel Swine-Origin Influenza A (H1N1) Virus in Humans. N Engl J Med. 2009 June 3. [Epub ahead of print].
 (11) Garten RJ, Davis CT, Russell CA, Shu B, Lindstrom S, Balish A, et al. Antigenic and Genetic Characteristics of Swine-Origin 2009 A(H1N1) Influenza Viruses Circulating in Humans. Science 2009 May 22. [Epub ahead of print]
(11) Garten RJ, Davis CT, Russell CA, Shu B, Lindstrom S, Balish A, et al. Antigenic and Genetic Characteristics of Swine-Origin 2009 A(H1N1) Influenza Viruses Circulating in Humans. Science 2009 May 22. [Epub ahead of print]
 (12) Centers for Disease Control and Prevention (CDC). Serum cross-reactive antibody response to a novel influenza A (H1N1) virus after vaccination with seasonal influenza vaccine. MMWR Morb Mortal Wkly Rep 2009;58(19):
(12) Centers for Disease Control and Prevention (CDC). Serum cross-reactive antibody response to a novel influenza A (H1N1) virus after vaccination with seasonal influenza vaccine. MMWR Morb Mortal Wkly Rep 2009;58(19):
521-4.
 (13) Centers for Disease Control and Prevention (CDC). Update: drug susceptibility of swine-origin influenza A (H1N1) viruses, April 2009. MMWR Morb Mortal Wkly Rep. 2009;58(16):
(13) Centers for Disease Control and Prevention (CDC). Update: drug susceptibility of swine-origin influenza A (H1N1) viruses, April 2009. MMWR Morb Mortal Wkly Rep. 2009;58(16):
433-5.
Remerciements
Laboratoire de premier niveau
Personnel technique des CNR : Frédérique Cuvelier, Vanessa Roca, Sébastien Le Gal, David Briand, Mathilde Benassaya.
Personnel technique de la CIBU : Christophe Batejat, Frédéric Fichenick, Claudine Rousseaux, Gilberte Coralie, Fabienne Lomprez, Yolande Arnoux.
Personnel d'astreinte de la CIBU : Ingrid Filliol, Alexandre Leclercq, Laurent Dacheux.
Personnel technique de la PF8 : Jean-Michel Thiberge, Coralie Tran, Julien Pujol.