Surveillance virologique en France : impact de la Covid-19 et perspectives
// Virological surveillance in France: The impact of COVID-19 and future prospects
Résumé
La surveillance épidémiologique est primordiale pour orienter les décideurs vers les mesures de gestion adaptées aux différents contextes d’une crise sanitaire. Dans le cadre de la pandémie liée au SARS-CoV-2, une surveillance épidémiologique a été mise en place, permettant notamment d’orienter la stratégie « tester, alerter, protéger » dans le but de freiner la propagation du virus.
Un système d’information de dépistage populationnel (SI-DEP) a été mis en place le 13 mai 2020 de manière très réactive, deux mois après la disponibilité des tests en mars 2020, pour recevoir systématiquement et d’une façon automatisée tous les résultats de tests (PCR, antigéniques) effectués quotidiennement par l’ensemble des laboratoires publics et privés, ainsi que les professionnels de santé habilités (France hexagonale et départements d’outre-mer).
Cet article décrit l’évolution de la surveillance virologique et la mise en place de SI-DEP pendant la crise sanitaire, depuis les phases de réflexions collaboratives entre les différents acteurs impliqués, le développement du système d’information et ses évolutions, jusqu’à l’exploitation et la diffusion des données et indicateurs produits notamment par Santé publique France.
Tout au long de la période de crise, plusieurs défis ont été rencontrés sur les plans scientifiques, techniques, règlementaires, ainsi que de forts enjeux de communication et de partenariats avec l’ensemble des parties prenantes. De fortes capacités d’adaptations, de cohésion et de résilience ont été nécessaires pour assurer l’ensemble des missions tout au long de cette période.
SI-DEP a montré l’importance d’avoir accès aux données des laboratoires de manière réactive pour assurer une surveillance épidémiologique efficace, et déclinée aux niveaux territoriaux les plus fins.
Abstract
Epidemiological surveillance provides decision-makers with essential information for implementing appropriate measures in response to a health crisis. In the context of the SARS-CoV-2 epidemic, epidemiological surveillance was set up as part of the strategy to trace, test and protect individuals to help control the spread of the virus.
When testing for SARS-CoV-2 became available in March 2020, the French information system for population-based screening (SI-DEP) was developed within 2 months and became operational on 13 May 2020. SI-DEP was designed to systematically and automatically receive the results of all tests (PCR, antigenic, etc.) performed daily by all public and private laboratories in mainland and overseas France.
This article describes the progression of virological surveillance and the implementation of SI-DEP during the health crisis, from the collaborative reflection phases involving various stakeholders to the development of the information system and its subsequent updates. We also explain how the data collected was exploited and communicated in the form of indicators produced by Santé publique France, the French national health agency.
The health crisis presented a number of scientific, technical and regulatory challenges in relation to surveillance. There were also high expectations in terms of communication and partnerships between stakeholders. A significant degree of adaptability, team cohesion and resilience was required to carry out the diverse tasks that this period demanded.
SI-DEP demonstrated the importance of real-time laboratory data to provide an exhaustive and continuous surveillance in conformity with epidemiological monitoring needs.
Introduction
L’apport des données de biologie médicale est essentiel à la surveillance des maladies infectieuses (MI) en France. En ce qui concerne les virus respiratoires, la surveillance virologique s’appuie sur le Centre national de référence des virus des infections respiratoires (CNR-VIR). L’objectif général de ce type de surveillance est de (i) détecter et isoler précocement les virus en circulation, (ii) d’en déterminer les caractéristiques antigéniques, et (iii) d’étudier leurs sensibilités aux antiviraux. Lors de l’émergence de la Covid-19, si le diagnostic était initialement basé sur des critères cliniques et radiologiques (scanner), une technique de diagnostic biologique fiable et sensible a été mise au point dès le 16 janvier 2020 par le CNR-VIR, afin de permettre la confirmation des cas, dont les trois premiers en France sont survenus le 24 janvier 2020 1.
À partir de début février 2020, et l’apparition des premières chaînes de transmission locales, il est devenu nécessaire d’augmenter le volume des tests diagnostiques et la capacité à pouvoir les traiter rapidement, afin de pouvoir faire face à l’augmentation de cas suspects. Dans un premier temps réalisés au CNR-VIR, puis dans un nombre croissant de laboratoires hospitaliers, le nombre de tests a rapidement augmenté avec l’arrivée sur le marché de kits de RT-PCR (polymerase chain reaction) ciblant le SARS-CoV-2, permettant un usage élargi à tous les laboratoires de biologie publics et privés (arrêté du 7 mars 2020) 2. C’est alors le début de la surveillance virologique à partir des données de biologie, qui va s’articuler autour du réseau 3-Labos, complété par la remontée des laboratoires hospitaliers en région.
Le réseau 3-Labos, mis en place par Santé publique France et qui fonctionne depuis 2012, est utilisé pour la surveillance de 21 maladies infectieuses (1), en particulier les arboviroses. Le réseau 3-Labos permet la remontée automatisée et quotidienne de données individuelles de trois laboratoires spécialisés privés (Eurofins-Biomnis, Cerba, et, depuis 2019, Inovie), qui centralisent les prélèvements que d’autres laboratoires leur sous-traitent et représentent une part importante de l’activité de biologie réalisée hors hôpitaux sur le territoire. Les objectifs de 3-Labos sont le suivi des tendances spatio-temporelles des MI ciblées et la contribution à l’investigation d’épidémies. Utilisé seul ou en complément d’autres dispositifs de surveillance, il est évolutif, donc particulièrement adapté en cas de maladie émergente pour obtenir des données de manière réactive.
Ainsi, la remontée des résultats vers Santé publique France via le système 3-Labos a permis de produire, dès le 14 mars 2020, l’un des premiers indicateurs de circulation du virus, le taux de positivité des tests RT-PCR pour SARS-CoV-2, et d’assurer son suivi dans le temps de façon quotidienne. Bien que ne couvrant qu’une partie des prélèvements réalisés sur le territoire, les données permettaient notamment de décrire les personnes testées et les personnes positives en termes d’âge, de sexe et de répartition géographique. En revanche, il ne permettait pas un suivi de la circulation virale à un niveau territorial fin et n’était pas adapté pour les activités de contact-tracing (recherche des contacts associés aux cas), compte tenu de sa couverture limitée et non exhaustive.
Ce constat, ainsi que la nécessité pour les pouvoirs publics de disposer d’un système permettant de mettre en lien les professionnels de santé, les personnes testées et les autorités de santé, a rapidement fait émerger le besoin d’un dispositif national centralisant de manière exhaustive, automatisée, systématique et réactive les données de l’ensemble des laboratoires publics et privés.
Cet article décrit l’évolution de la surveillance virologique opérée par Santé publique France pendant la crise sanitaire causée par le SARS-CoV-2, depuis les phases de réflexions collaboratives en février 2020 entre les différents acteurs impliqués, le montage du système d’information et ses évolutions, l’exploitation des données recueillies pour la production d’indicateurs de surveillance, puis la transition vers d’autres systèmes d’information à partir de juillet 2023. Seront également abordés les défis liés aux évolutions successives de ce système pour s’adapter à la pandémie, ainsi que les enjeux liés à la diffusion de données de manière réactive, notamment via l’open data.
Retour d’expérience du système d’information de dépistage populationnel (SI-DEP)
La mise en place d’un nouveau système d’information : SI-DEP
Le régime de l’état d’urgence sanitaire créé par la loi n° 2020-290 du 23 mars 2020 d’urgence pour faire face à l’épidémie de SARS-CoV-2 3 a permis de prendre les mesures rendues nécessaires par ces circonstances, dont la création de systèmes d’information nationaux pour la remontée de données de santé. Le Système d’information de dépistage populationnel (SI-DEP) est un système d’information (SI) national sécurisé pour le recueil des résultats de tests de dépistage de SARS-CoV-2 effectués en France.
Mis en production le 13 mai 2020, SI-DEP a été construit dans l’objectif de répondre aux besoins de surveillance épidémiologique à partir des données de biologie pendant la crise sanitaire, et afin d’orienter les mesures de gestion permettant de limiter l’impact de l’épidémie, d’alimenter les outils de contact-tracing, de permettre l’accès à des données anonymisées pour la recherche (décret n° 2020-551 du 12 mai 2020 4), et de constituer un outil d’échange entre les professionnels de santé et les citoyens.
Ce SI est né d’un partenariat entre le ministère de la Santé et de la Prévention, via la Délégation du numérique en santé et la Direction générale de la santé (responsable de traitement et maître d’ouvrage), l’Assistance publique-Hôpitaux de Paris (AP-HP, maître d’œuvre et chef de file d’un consortium), les laboratoires de biologie médicale publics et privés ainsi que leurs prestataires en SI, l’Agence du numérique en santé (ANS), Santé publique France, la Direction de la recherche, des études, de l’évaluation et des statistiques (Drees), la Caisse nationale d’assurance maladie (Cnam), et les agences régionales de santé (ARS).
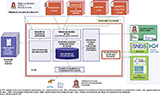
SI-DEP a permis le raccordement de près de 5 000 laboratoires privés et publics disposant ou non d’automates pour la réalisation simultanée d’un grand nombre de tests (dits « MGI »), dont 600 plateaux techniques, à un système d’information centralisé pour le recueil des tests RT-PCR. Les laboratoires poursuivent la saisie des données concernant les résultats de tests et les données sociodémographiques dans leur système de gestion de laboratoire (SGL), qui sont automatiquement transmises vers SI-DEP. Les données saisies sont collectées dans une base de donnée nationale qui alimente les différents acteurs pour les missions dont ils ont la charge. Les données nominatives sont transmises à la Cnam et aux ARS pour les missions de contact-tracing. Seules les données pseudonymisées sont transmises à Santé publique France et à la Drees pour le suivi épidémiologique, et au Système national des données de santé (SNDS) pour la recherche (figure 1). SI-DEP a également facilité l’échange des résultats de tests entre les professionnels de santé et les patients, ainsi que la génération de certificats de tests avec des QR codes pour les passes sanitaires pendant les périodes de confinement et mesures de restrictions nationales.
Un système d’information en constante évolution
Afin de permettre une surveillance capable de s’adapter en permanence à l’épidémie, plusieurs évolutions majeures de SI-DEP ont été nécessaires. En effet, lors de sa mise en place en mai 2020, SI-DEP permettait uniquement la remontée des résultats de RT-PCR pour le dépistage du SARS-CoV-2. Cependant, au cours de la pandémie, la stratégie de tests a évolué de sorte à être toujours plus réactive pour mettre en place de nouvelles mesures de gestion de l’épidémie.
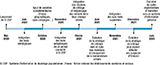
Les changements majeurs ont concerné l’intégration successive : des résultats de tests sérologiques en juillet 2020 ; des résultats de tests antigéniques (TAG) effectués par différents professionnels de santé (pharmaciens, médecins, masseurs-kinésithérapeutes, infirmiers, chirurgiens-dentistes ou encore sages-femmes) en novembre 2020 ; des résultats de criblage et de séquençage pour le suivi des variants en février 2021 puis de certaines mutations ; ainsi que des résultats des autotests supervisés (figure 2).
L’intégration des résultats des TAG dans SI-DEP a notamment pu être effectuée à travers la mise en place d’un portail de saisie connecté à SI-DEP pour les professionnels de santé concernés. Pour faciliter l’exploitation des données saisies manuellement, des contrôles qualité ont été mis en place, afin de garantir une qualité des données comparable à celles remontées par les laboratoires.
Le volume des tests RT-PCR et TAG saisis quotidiennement et la stratégie de dépistage ont aussi évolué au cours du temps, nécessitant une adaptation continue de l’infrastructure informatique de SI-DEP et une augmentation de ses capacités machine afin de pouvoir absorber la volumétrie croissante de tests réalisés, sans engendrer de problème de transmission de données entre les différentes parties concernées. Lors du lancement de SI-DEP, 40 000 tests par jour étaient enregistrés dans la base. Ce volume a augmenté à 500 000 en mars 2021, puis 1 million en août 2021 pour atteindre jusqu’à 2,7 millions de tests par jour en janvier 2022 (2), nécessitant une adéquation du dimensionnement des infrastructures techniques tout au long de la crise (la capacité maximale de SI-DEP ayant été portée à 4 millions de tests par jour).
La construction des indicateurs de surveillance virologique
Les flux de données en temps réel issus de l’ensemble des laboratoires et des autres professionnels de santé vers SI-DEP ont rendu possible la production quotidienne d’un ensemble d’indicateurs épidémiologiques déclinés à un niveau territorial très fin, qui ont permis de décrire l’évolution spatio-temporelle de l’épidémie. Le taux d’incidence (nombre de personnes nouvellement testées positives rapporté à la taille de la population), le taux de positivité (nombre de personnes nouvellement testées positives rapporté au nombre total de personnes testées) et le taux de dépistage (nombre de personnes testées rapporté à la taille de la population) sont les indicateurs clés calculés à partir des données issues de SI-DEP (3) (figure 3 et 4). Ces indicateurs sont notamment basés sur les résultats des différents tests biologiques (RT-PCR, TAG) et les caractéristiques individuelles des personnes (âge, sexe, lieu de résidence, présence de symptômes, etc.).
Le calcul des patients incidents – au regard de la définition de cas retenue, selon les recommandations de la Haute Autorité de santé (HAS), du Haut Conseil de la santé publique (HCSP), du Centre européen de prévention et de contrôle des maladies (ECDC) et de l’Organisation mondiale de la santé (OMS), qui décrit un délai de 60 jours par rapport à une précédente infection – a été rendu possible grâce au principe de pseudonymisation. Les informations nominatives d’une personne (nom, prénom, date de naissance et sexe) sont utilisées dans un algorithme qui permet de produire un code unique (pseudonyme) pour chaque personne, donc de repérer des infections successives pour une personne donnée.
Ce pseudonyme contribue à la sécurisation et l’anonymisation des données transmises aux autorités sanitaires, et permettent également d’effectuer des appariements entre les différentes bases de données en lien avec le SARS-CoV-2, comme par exemple les bases de données de vaccination (VAC-SI) et d’hospitalisation (SI-VIC).
Lors de la collecte des informations individuelles du patient au moment de la réalisation d’un test Covid-19, le laboratoire recueille l’adresse de la personne (numéro, voie, code postal et nom de la localité de résidence). Cette adresse complète fait l’objet d’un processus de géocodage (affectation de coordonnées géographiques longitude et latitude) utilisé pour calculer l’Iris (îlot regroupé pour l’information statistique – brique de base en matière de diffusion de données infra-communales 5) de résidence de la personne testée. Ce processus a permis de décliner des indicateurs à des échelons géographiques très fins (Iris, commune, établissements publics de coopération intercommunale – EPCI), et contribue à l’anonymisation des données envoyées aux autorités sanitaires. La précision de ces indicateurs a permis de décrire la dynamique de l’épidémie et de constituer une aide à la décision par les autorités locales, afin d’adapter les mesures de gestion au plus près des territoires.
 Agrandir l'image
Agrandir l'image Agrandir l'image
Agrandir l'imageL’utilisation des indicateurs issus de SI-DEP pour la gestion de l’épidémie
Les indicateurs issus de SI-DEP en complément des autres sources de données ont contribué à la prise de décision et à la mise en place de mesures de gestion pour faire face à la propagation du virus. Les seuils d’alerte sur le taux d’incidence par exemple, définis pour le suivi de l’évolution de la circulation virale, ont également été utiles pour la déclinaison des différentes mesures sanitaires par les pouvoirs publics (confinement, port du masque, couvre-feu, vaccination…). Ils ont contribué à l’identification des différents pics de l’épidémie, ainsi que des périodes de recrudescence des cas, et à l’adaptation des mesures de freinage, en fonction de la situation sanitaire dans un territoire donné 6. Par exemple, en octobre 2020, lors de la deuxième vague de Covid-19 et à la suite de l’analyse des indicateurs issus de l’ensemble des sources de surveillance, un couvre-feu a été instauré dans plusieurs métropoles pour freiner la propagation du virus. À la suite de la dégradation de la situation sanitaire sur tout le territoire, cette mesure a évolué vers un confinement national de fin octobre jusqu’à mi-décembre 2020 6.
En début d’année 2021, à la suite de l’émergence de variants du SARS-CoV-2, la mise en œuvre de RT-PCR de criblage dans les laboratoires a permis de détecter certaines des mutations caractéristiques et d’assurer le suivi de leur diffusion. En complément, les capacités de séquençage ont été progressivement renforcées via le consortium pour la surveillance et la recherche sur les infections à pathogènes émergents via la génomique microbienne (Emergen), coordonné par Santé publique France et l’ANRS – Maladies infectieuses émergentes (ANRS-MIE) (4). Afin de suivre au niveau national les détections de certains variants connus et considérés comme ayant un impact important en santé publique (plus grande transmissibilité, échappement immunitaire…), le SI-DEP s’est adapté en incluant de nouvelles données saisies par les laboratoires : un premier champ « joker » pour le suivi des résultats de RT-PCR de criblage et un second pour le suivi des résultats de séquençage. L’exploitation de ces champs a permis de produire de nouveaux indicateurs épidémiologiques (taux de criblage, taux de détection d’une mutation donnée) et une estimation très réactive de la proportion de variants porteurs de ces mutations. Elle a aussi permis, dans les premières semaines d’introduction de ces variants, de mettre en place des actions spécifiques de contact-tracing dans l’objectif de contenir leur diffusion.
Alors qu’en début d’année 2021, seuls les variants Alpha, Beta et Gamma étaient classés comme préoccupants 7, le nombre croissant de variants a nécessité, en fin d’année, l’évolution de l’exploitation des résultats des RT-PCR de criblage vers un suivi des mutations d’intérêt, pour pouvoir suivre l’évolution spatio-temporelle de la proportion des infections dues à un virus porteur de ces mutations.
Les indicateurs virologiques ont également permis de suivre la propagation du SARS-CoV-2 dans certaines populations spécifiques. À la suite de la mise en place de la stratégie de dépistage réactif en milieu scolaire en automne 2021, les indicateurs issus de SI-DEP ont ainsi été déclinés par classe d’âge représentant les différents cycles scolaires (0-2 ans, 3-5 ans, 6-10 ans, 11-14 ans, 15-17 ans, 18 ans et plus) (5) Ils ont favorisé la mise en place d’aménagements spécifiques en fonction du taux d’incidence chez les élèves pour éviter la fermeture systématique des classes en présence de cas probable ou confirmé 8.
Les défis techniques et la sécurité des systèmes d’information
La connexion à SI-DEP de l’ensemble des laboratoires réalisant des tests de dépistage du SARS-CoV-2 dans des délais très contraints, entre les mois de mars et mai 2020, a été source de nombreux défis techniques.
Afin de mettre en place une base de données centralisée qui permette la réception de l’ensemble des résultats de tests issus des laboratoires privés (plus de 4 100 laboratoires) et publics (plus de 400 laboratoires), un important travail d’homogénéisation et d’interopérabilité entre les différents systèmes a été conduit.
Ce travail mené par le ministère de la Santé et de la Prévention et l’AP-HP, en lien avec les éditeurs de systèmes d’information de laboratoire et Interop’Santé, a notamment conduit à des développements pour l’utilisation de normes d’échange (HL7 ; H’) et référentiels LOINC (Logical Observation Identifiers Names & Codes) qui s’appuient sur une terminologie de référence internationale pour la codification des résultats d’examen de biologie dans le but de standardiser les informations envoyées par les laboratoires et de faciliter l’exploitation des bases de données.
Ce travail d’adaptation des infrastructures a dû être effectué en miroir, et la synchronisation à la fois par l’AP-HP pour la centralisation des données et l’envoi vers les partenaires avals, et par Santé publique France pour la réception des données anonymisées, et ce à plusieurs reprises. Un important dispositif de monitoring des flux de données a dû être mis en place, afin de détecter et résoudre toute anomalie de transmission de manière très réactive.
Le recueil de données de santé individuelles impose un contexte de sécurité informatique et de protection des données très importantes. La mise en œuvre de la collecte et du traitement des données s’est effectuée dans le respect des principes posés par le Règlement général sur la protection des données (RGPD) et de la loi informatique et libertés. Une homologation au référentiel général de sécurité, une certification hébergeur de données de santé, et des analyses d’impact relatives à la protection des données ont notamment été mises en place par l’ensemble des parties prenantes pour garantir la sécurité et la protection des données. L’AP-HP a renforcé la sécurisation de SI-DEP avec des outils de cyberattaque du fait de risques accrus liés à ce SI stratégique et national. La Commission nationale de l’informatique et des libertés (Cnil) a également diligenté de nombreux contrôles sur pièces et sur place du SI-DEP, et a rendu des avis favorables aux évolutions du décret afférent pris en conseil d’État.
Les enjeux scientifiques et de santé publique
La surveillance virologique a évolué tout au long de la crise, en accord avec les pratiques de test, l’évolution du SARS-CoV-2 et l’émergence de variants et mutations, et la mise en place de mesures de gestion.
L’émergence des variants fin 2020 a représenté un défi majeur. Le choix de la nomenclature à adopter (Pangolin, Nexstrain, GISAID – Global Initiative on Sharing Avian Influenza Data –, …) a nécessité de nombreuses discussions entre le CNR-VIR, les partenaires, ainsi que les laboratoires et les éditeurs de systèmes d’information des laboratoires (SIL), impliquant des délais importants pour prise en compte dans SI-DEP. Les difficultés liées à une intégration tardive dans le système d’information, la complétude du champ manuel par les laboratoires et les règles de protection des données spécifiques en génétiques n’ont pas permis d’exploiter correctement cette information, en raison d’un manque d’exhaustivité et de qualité des données saisies. La mise en place du projet Emergen a permis de suivre l’évolution génétique du SARS-CoV-2 pour détecter l’émergence et la distribution spatio-temporelle de variants.
Le changement de la stratégie de criblage d’une recherche de variant à une recherche de mutation fin 2020 a été accompagnée par une nouvelle nomenclature à adopter, ce qui a impacté la remontée des informations dans SI-DEP. Cela a nécessité une nouvelle phase d’adaptation par les laboratoires pour assurer une remontée des informations à un niveau national.
Santé publique France a construit des indicateurs épidémiologiques d’une forte précision pour suivre au mieux l’épidémie. La production quotidienne d’indicateurs permettant le suivi de l’épidémie à des échelons géographiques infra-départementaux (communes, EPCI, Iris), et pour différentes classes d’âges, a représenté un enjeu majeur pour la continuité d’activité.
À la précision des indicateurs calculés pour décrire au mieux la population, s’ajoute l’enjeu d’une production quotidienne d’un grand nombre d’informations selon des formats différents (fichiers de données, bulletins épidémiologiques, tableaux de bord, etc.), ce qui a nécessité la mise en place de solutions innovantes pour l’automatisation et le monitoring des traitements, ainsi qu’une très importante mobilisation humaine de l’ensemble des acteurs.
L’importance de ces données pour le suivi de la pandémie a imposé de fortes contraintes pour garantir l’opérationnalité du dispositif sans discontinuité, nécessitant par exemple la mise en place d’astreintes de soir et week-ends pour la mise en relation de l’ensemble des acteurs impliqués dans le dispositif, depuis la collecte des données, la centralisation et leur stockage, l’envoi et le traitement par les agences sanitaires. Des modes dégradés de transmission des données ont pu être développés pour pallier des anomalies ponctuelles.
Les enjeux de partenariat et de communication
La collaboration rapprochée entre les acteurs institutionnels et de terrain a été un enjeu majeur dans la mise en place, l’évolution et la maintenance d’un tel dispositif. Le développement et les évolutions successives du système ont nécessité une mobilisation de tous les instants de l’ensemble des acteurs impliqués, depuis les professionnels de santé des secteurs publics et privés du fait de leurs pratiques, les éditeurs de solution informatique, l’intégration des évolutions par l’AP-HP, jusqu’aux agences sanitaires pour le recueil des données et la restitution d’indicateurs épidémiologiques, le tout sous le pilotage du ministère de la Santé et de la Prévention.
Dans ses missions de mise à disposition d’indicateurs épidémiologiques, Santé publique France a été conduit à communiquer auprès d’un large public, dans l’objectif d’accompagner les évolutions du dispositif. De nombreuses notes méthodologiques ont été rédigées à destination des autorités sanitaires, des partenaires, de la presse, mais également auprès du grand public.
La publication de ces informations en open data a nécessité une pédagogie importante au regard des indicateurs produits, et devait être accompagnée par une communication détaillée pour être compréhensible et réutilisable par le grand public. Des séances de décryptages hebdomadaires ont été organisées par le service de la communication de Santé publique France pour répondre aux différentes interrogations des journalistes et présenter les évolutions du dispositif en toute transparence. Ces indicateurs ont pu être repris par de nombreuses personnes, professionnelles ou non, et ont permis l’alimentation de nombreux tableaux de bord et outils de visualisation (TousAntiCovid ou CovidTracker (6) par exemple).
Conclusion
Les expériences internationales et leçons tirées
D’autres expériences similaires ont été observées à l’échelle européenne pour suivre l’évolution de la pandémie. La flexibilité et l’adaptabilité des systèmes d’information pour la surveillance virologique ainsi que les réseaux préexistants entre les laboratoires, les professionnels de santé et les acteurs de santé publique ont facilité cette transition, et renforcé la coopération entre eux 9. Face à cette crise, plusieurs pays européens ont adapté les systèmes de surveillance virologique déjà en place pour pouvoir suivre la propagation du virus à des niveaux très fins, collecter des données en temps réel, mettre en place des mesures de prévention et suivre leurs impacts 9.
Tout comme la France, l’Allemagne a adapté son système de surveillance déjà existant (pour le suivi de la résistance aux antimicrobiens) pour recevoir les résultats de tests de SARS-CoV-2 effectués en laboratoires de manière automatique et suivre l’épidémie en temps réel. Ce système couvre l’ensemble des laboratoires sur le territoire qui traitent et analysent les échantillons provenant des hôpitaux, centres de soins primaires et services ambulatoires 10. La Belgique a également mis en place un système de surveillance exhaustif comparable à la France qui a permis de récolter les données de tests de laboratoires quotidiennement, en passant dans un premier temps par une centralisation des résultats via le réseau de laboratoires sentinelles belges, suivie par la mise en place d’une base de donnée centralisée pour la remontée des résultats directement à partir des SI des laboratoires 11. L’Espagne a adapté son système de surveillance électronique (SiViES) pour inclure la surveillance du SARS-CoV-2 9.
Si les dispositifs de collecte de données étaient comparables, les définitions de cas et les modalités de calcul des indicateurs épidémiologiques pouvaient différer d’un pays à l’autre, conduisant à des difficultés dans les comparatifs des tendances entre les pays. SI-DEP a un caractère exceptionnel dans son périmètre de fonctionnalité en comparaison aux autres systèmes européens, notamment au travers de fonctionnalités tournées vers les citoyens pour remettre des certificats numériques à tous les patients dans le cadre de la mise en place du passe sanitaire.
Le partage de ces expériences permet notamment aux autorités telles que l’ECDC de produire des recommandations sur les évolutions à développer et maintenir pour faire face à de futures pandémies, comme la mise en place de systèmes de surveillance intégrés et représentatifs pour le suivi des infections respiratoires 12.
Les perspectives de SI-DEP
La pandémie de Covid-19 a démontré l’importance d’avoir accès à des données de qualité de manière réactive pour pouvoir mettre en place des actions adaptées dans un contexte de crise sanitaire 13. Les investissements effectués dans le champ du numérique en santé en France depuis plus de 10 ans comme la mise en place de la carte e-CPS, France Connect, du système Sesam-Vitale, etc., et l’usage généralisé d’outils numériques via les téléphones ou les PC a facilité l’utilisation et l’exploitation de la donnée dans ce contexte particulier.
L’utilisation de formats d’échanges standardisés et des référentiels internationaux permettent d’accélérer la mise en place de ces systèmes, et d’optimiser les échanges et l’interopérabilité des systèmes à des échelles nationales et internationales.
La mise en place de systèmes de surveillance exhaustifs permettant de répondre à plusieurs objectifs a démontré la nécessité de pouvoir traiter une quantité massive d’informations et de disposer d’infrastructures techniques robustes et prêtes à collecter et absorber des données en grande quantité de manière réactive (en temps de crise ou non). Ce besoin a mis en lumière l’importance des métiers de l’informatique pour la mise en place, la maintenance et l’évolution de tels systèmes, et de la data science pour l’exploitation massive et automatisée des données.
La pandémie liée au SARS-CoV-2 a mis en avant le besoin en santé publique de s’appuyer sur des systèmes d’information nationaux et exhaustifs, notamment à partir des données de biologie médicale pour le suivi de nombreuses maladies. L’avenir des systèmes de surveillance repose ainsi notamment sur l’exploitation plus systématique des données de santé générées par les professionnels de santé et déjà disponibles au sein de bases de données structurées, et sur leur diffusion via des SI modernes et automatisés, permettant d’alléger la charge de travail des déclarants, tout en garantissant la sécurité des données des patients.
Anticiper la mise en place de tels dispositifs est capital, afin de renforcer les surveillances existantes et d’anticiper la survenue future d’événements pour lesquels il sera nécessaire de disposer de données de manière réactive et exhaustive.
Le système SI-DEP pourra servir d’expérience et de modèle dans le cadre de la mise en place de futurs systèmes de surveillance intégrés, étendus à d’autres pathologies, ne se limitant pas aux maladies infectieuses.
La publication du décret n° 2023-700 du 31 juillet 2023 14 et de l’arrêté du 7 août 2023 15 relatif à la transmission obligatoire de données individuelles à l’autorité sanitaire et à la création du traitement de données à caractère personnel « LABOé-SI » garantit la remontée des résultats des laboratoires publics et privés, et permettra la pérennisation de la surveillance virologique pour la Covid-19, mais également l’intégration progressive d’autres maladies. Ce système aura vocation à être utilisé à la fois pour les surveillances pérennes et pour la gestion de crise sanitaire exceptionnelle via un module de crise qui permettra la mobilisation d’autres professionnels de santé.
Remerciements
Les auteurs remercient Corinne Danan (venue en renfort de l’Anses à Santé publique France de mars à mai 2020) pour sa contribution aux travaux préparatoires à la mise en place de l’outil SI-DEP, tous les acteurs publics et privés ayant contribué à la création de cet outil, et tous les laboratoires de biologie médicale et professionnels de santé ayant contribué à l’alimentation de cette base de données nationale.
Liens d’intérêt
Les auteurs déclarent ne pas avoir de liens d’intérêt au regard du contenu de l’article.
Références
Citer cet article
fr/#c=indicator&i=sp_capa_quot.
t&s=2022-05-01&t=a01&view=map2
fr/#c=indicator&i=sp_7j_nt.tx_ti_
gliss&s=2023-08-23-2023-08-29&t=a01&view=map2
fr/#c=indicator&f=02&i=sp_7j_cage_
scol.ti&s=2023-06-21-2023-06-27&t=a01&view=map2