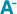Expérimentation d’un transfert automatisé des données microbiologiques de laboratoires de biologie médicale de ville à des fins de surveillance épidémiologique, réseau Labville, France, 2005-2009
// Experiment of an automated system of microbiological data collection in private-sector, community-based medical laboratories for epidemiological surveillance, Labville network, France, 2005 -2009
Résumé
L’expérimentation Labville, conduite de 2005 à 2009 pour contribuer à surveiller la résistance aux antibiotiques (RATB), mettait en œuvre un transfert automatisé, quotidien et sécurisé des données de bactériologie d’un réseau de 69 laboratoires de ville (LABM). Les données étaient capturées par reconnaissance des flux d’impression des comptes-rendus d’analyse.
L’évaluation du système a considéré le nombre de LABM équipés et les indicateurs épidémiologiques produits.
Entre 2005 et 2009, 44 LABM ont transmis leurs données. Les proportions de résistance produites dans chaque espèce bactérienne étaient cohérentes avec la littérature : 20,9% des souches de Staphylococcus aureus résistantes à la méticilline et 2,2% des souches d’Escherichia coli résistantes aux céphalosporines de 3e génération. L’instabilité des formats d’impression invalidait toutefois régulièrement la récupération des données. Les règles d’extraction et de transcodage des données devaient être adaptées constamment, motivant l’arrêt de l’expérimentation en 2009.
L’expérimentation Labville a confirmé que les LABM disposaient des données pertinentes pour surveiller la RATB en ville, mais n’a pas validé de solution technique pérenne pour les récupérer. La surveillance de la RATB en ville reste à renforcer, notamment face à la diffusion des souches d’Escherichia coli productrices de bêtalactamases à spectre étendu. Plusieurs alternatives peuvent y contribuer : études ciblées (possiblement répétées), production d’indicateurs propres à la médecine de ville par les Centres nationaux de référence et collaboration avec les réseaux existants.
Abstract
The Labville experiment, carried out from 2005 to 2009 to contribute to antimicrobial resistance (AMR) surveillance, was an automated system of microbiological data collection based on the recognition of printer-flow and internet transmission from 69 private-sector, community-based medical laboratories (CBML).
The system was evaluated according to the number of CBML transmitting data and to the values of produced epidemiological indicators.
Between 2005 and 2009, 44 labs transmitted data. The proportions of resistance within species were consistent with the literature: 20.9% of Staphylococcus aureus strains were resistant to methicillin and 2.2% of Escherichia coli strains were resistant to 3rd generation cephalosporins. However, data were regularly lost because of a high instability of printing formats, requiring the constant monitoring of connected laboratories and the adaptation of rules for printer-flow reading or data encoding. This led to end the Labville experimentation in 2009.
Labville confirmed that relevant data for AMR surveillance in the community are available in CBML. However, a sustainable solution was not identified to collect them. The strengthening of AMR surveillance in the community remains important, especially considering the spread of ESBL-producing Escherichia coli. Alternative solutions should be explored in order to produce community-based indicators, such as conducting targeted studies (possibly repeated) or collaborating with national reference centres or existing surveillance networks.
Introduction
La consommation des antibiotiques crée une pression de sélection favorisant l’apparition et la dissémination de bactéries résistantes aux antibiotiques (RATB) 1. Surveiller la RATB est important pour guider les mesures permettant d’en maîtriser la diffusion 2. En France, 80% des antibiotiques sont prescrits en ville 3. Cependant, la surveillance de la RATB est principalement basée sur des laboratoires d’établissements de santé publics ou privés 4 et n’est pas restreinte aux laboratoires de biologie médicale de ville (LABM) 5. En ville, elle reste à renforcer. Seuls quelques réseaux fédérés au sein de l’Observatoire national de l’épidémiologie de la résistance bactérienne aux antibiotiques (Onerba) - Réseau Aquitaine, Epiville ou AforcopiBio - produisent, de manière discontinue, certaines données spécifiques aux LABM.
L’Institut de veille sanitaire (InVS) a donc choisi, en 2000, de créer un réseau de LABM, sélectionnés aléatoirement pour représenter 5% des examen cytobactériologiques des urines (ECBU) réalisés en France métropolitaine, afin de surveiller en continu la RATB des infections diagnostiquées en ville, sans restriction à des couples bactéries-antibiotiques cibles et avec une représentativité nationale 6. L’objectif de ce réseau Labville était d'automatiser la transmission des données de bactériologie des LABM vers l’InVS, après une étude de faisabilité conduite en 2003 7. Cet article décrit les résultats issus de l’expérimentation du système Labville, qui s’est déroulée de 2005 à 2009 après identification d’un prestataire pour la réalisation du système automatisé, dans le cadre d’un marché public.
Méthodes
Solution retenue
En raison du faible nombre de réponses à l’appel d’offres d’éditeurs développant des systèmes d’information de laboratoire (SIL, figure 1), l’InVS a choisi d’équiper les 69 LABM du réseau Labville d’une solution innovante qui contournait ces SIL en captant les données dans le flux d’impression des comptes rendus de bactériologie 6.
 Agrandir l'image
Agrandir l'image
Les stratégies de lecture des flux d’impression étaient définies par LABM et par type de prélèvements. Les fichiers générés étaient transmis à l’InVS par connexion Internet sécurisée (figure 2).
La validation technique s’effectuait en deux temps : d’abord sur un jeu d’analyses-type, puis sur une période de quatre mois considérant la réception quotidienne des données, l’exhaustivité (>90%) des comptes rendus reçus et leur complétude. Le traitement quotidien des fichiers reçus alimentait une base de données individuelles (accord Cnil N°05-1008).
Les fichiers reçus n’étant pas structurés comme initialement prévu au cahier des charges, deux traitements s’imposaient avant exploitation des données : un transcodage et le rapprochement des résultats de culture et d'antibiogramme, reçus séparément et sans identifiant commun pour un même résultat d’analyse. Pour ce rapprochement, la concordance du type de prélèvement et de bactérie entre résultat(s) et antibiogramme(s) d’un même compte-rendu, le nombre de résultats, le nombre d’antibiogrammes et le nombre de bactéries dans un même résultat étaient notamment pris en compte.
Après traitement, les variables disponibles dans la base de données étaient l’âge, le sexe, le département de résidence, le statut du patient (hospitalisé ou non), un identifiant patient unique, le numéro de dossier, la nature (urines, peau et phanères, pus fermé ou non...) et la date du prélèvement, le résultat de la culture et le résultat interprété de chaque antibiotique testé.
Évaluation
La performance du système Labville a été évaluée au regard a) du nombre de LABM ayant satisfait aux conditions de l’installation du système et b) des indicateurs de surveillance produits pour cinq espèces bactériennes (Escherichia coli, Staphylococcus aureus, Campylobacter, Salmonella, Streptococcus pneumoniae) sur les résultats reçus au second semestre 2008, période où les programmes de lecture des flux d’impression étaient optimaux.
Analyse épidémiologique
Les doublons épidémiologiques sur la période d’étude ont été exclus. Les souches retenues étaient :
- la première souche de l’espèce étudiée isolée chez un même patient, pour l’expression des résultats, tous sites de prélèvement confondus ;
- la première souche de l’espèce étudiée isolée d’un même type de prélèvement et chez un même patient, pour l’expression des résultats par site de prélèvement ;
- la souche isolée du prélèvement de plus grande valeur clinique en cas d’isolement d’une même souche à partir de plusieurs prélèvements réalisés le même jour. Les critères de valeur cliniques retenus étaient ceux habituellement utilisés par les microbiologistes et les cliniciens considérant la stérilité du produit prélevé et le risque de contamination au cours du prélèvement.
Les doublons d’impression étaient par ailleurs exclus sur la base de six variables (numéro de patient, numéro de dossier, date et heure de prélèvement, nature et site).
La nature des prélèvements et celle des bactéries isolées ont été décrites. Des proportions de résistance dans l’espèce ont été calculées. Les résultats interprétés de sensibilité aux antibiotiques (sensible, intermédiaire ou résistant) étaient ceux validés par les biologistes. L’analyse épidémiologique a été conduite avec le logiciel SAS® (version 9.1, SAS Institute Inc., Cary, NC, USA).
Résultats
Le système Labville a été validé pour 44 des 69 LABM du réseau. Ceux-ci ont transmis des données sur des durées variables, de 7 mois à 2 ans selon le LABM. Ils couvraient 3,4% de l’activité nationale de bactériologie de ville, mesurée en nombre d’ECBU réalisés. Au total, d’octobre 2005 à avril 2009, 753 632 comptes rendus sont parvenus à l’InVS (figure 3).
 Agrandir l'image
Agrandir l'image
Concernant l’installation du système, 3 LABM n’ont pas été équipés du boîtier informatique nécessaire. Parmi les 66 LABM pourvus d’un boîtier, 5 ont eu une stratégie de lecture validée sur un jeu d’analyses-type sans modification ; de un à quatre réajustements par le prestataire ont été nécessaires pour valider celles de 55 autres LABM, et 6 LABM n'ont pas eu de stratégie proposée par le prestataire en raison de difficultés techniques insurmontables lors de l’installation des boîtiers. Cinq versions successives du programme de traitement du flux d’impression ont été installées dans les LABM équipés. Au total, 60 LABM ont satisfait à la validation initiale des stratégies de lecture et 44 à la validation technique complète.
En avril 2009, 36 LABM restaient fonctionnels parmi les 44 LABM pour lesquels le système Labville avait été validé. Parmi eux, 7 avaient connu une seule interruption de transmission de courte durée (inférieure à 20 jours) et 7 n’en avaient jamais connu. Concernant les 33 autres LABM non fonctionnels, 8 ne transmettaient plus de données après une période moyenne de 9 mois et 25 n’avaient pas atteint le stade du fonctionnement en routine. Les installations inachevées relevaient de stratégies de lecture non proposées, de déménagements du LABM ou du retard du prestataire sur le projet (n=5), de transmissions de données non activées par défaut de traitement du flux d’impression (n=2) ou de transmissions restées insatisfaisantes (n=14). Concernant le fonctionnement en routine du système, parmi les 29 interruptions de réception intervenues entre janvier et septembre 2008, 11 pannes d’imprimante et 5 débranchements inopinés de boîtier ont été résolus après l’identification d’un dysfonctionnement par l’InVS et 13 autres ont été résolus par le prestataire dans un délai variant de 6 à 70 jours.
Les indicateurs épidémiologiques produits au cours de la période d’évaluation (juillet à décembre 2008) portent sur 79 930 résultats de bactériologie dédoublonnés, issus d’environ 120 000 résultats reçus à l’InVS. La majorité des résultats reçus concernait des prélèvements urinaires (74,8%). Un tiers des résultats étaient des cultures positives. Au total, 28 087 bactéries étaient identifiées ; les plus fréquentes étaient les entérobactéries (53,8%), principalement E. coli (41,7%), suivi des cocci à Gram positif (31,2%).
E. coli était surtout isolé de prélèvements d’urine (91,1%) et fréquemment testé pour céfotaxime, ceftriaxone ou ceftazidime (93,3%). Parmi les souches isolées d’urines et testées pour ces antibiotiques, 2,2% étaient résistantes et 1% de sensibilité intermédiaire. S. aureus était isolé de prélèvements de peau et phanères (30,9%), d’urine (14,3%) et de pus non fermé (7,8%) ou fermé (1,2%). La majorité des souches avait été testée pour la méticilline (85,1%). Parmi les souches isolées de pus et testées pour cet antibiotique, 20,3% étaient résistantes ; respectivement 19% et 35,5% étaient résistantes parmi celles isolées de prélèvement de peau et phanères, ou d’urines. Le nombre de souches isolées pour les trois autres bactéries cibles retenues (Salmonella (n=104), Campylobacter (n=95) et Streptococcus pneumoniae (n=38)) était insuffisant pour interpréter les proportions de résistance chez ces espèces.
Discussion
L’expérimentation Labville a montré qu’il était possible de transmettre quotidiennement à l’InVS, sous un format unique et de façon automatisée, les données de plusieurs dizaines de LABM. Néanmoins, la solution technique retenue comme alternative à l’extraction directe des données des SIL – innovante, ambitieuse mais très instable – n’a été validée que pour 44 (64%) des 69 LABM du réseau. Elle n’a pas permis de générer des indicateurs de manière pérenne et d’assurer une rétro-information régulière des laboratoires participants. L’importante charge de travail nécessaire à l’équipement et au maintien dans le temps de cette transmission automatisée a conduit l’InVS à interrompre cette expérimentation fin novembre 2009.
Les données reçues ont toutefois permis de décrire l’activité de bactériologie des LABM équipés et de calculer des proportions de résistance au sein des principales espèces bactériennes isolées, globalement cohérentes avec les données de la littérature française : prédominance des prélèvements urinaires 7,8, prédominance des E. coli 9,10 et proportions de résistance 10,11,12. La portée de ces résultats peut toutefois être limitée en l’absence de démarche de contrôle qualité intégrée à l’expérimentation Labville, alors que la littérature décrit son intérêt 11. Enfin, seuls deux des cinq indicateurs cibles retenus pour l’évaluation épidémiologique du système ont pu être calculés, questionnant la pertinence du périmètre de recueil de données initialement retenu (toutes bactéries).
La solution informatique retenue reposait sur la capture des informations utiles à partir du flux d’impression des comptes rendus, l’InVS s’affranchissant ainsi des contraintes liées aux éditeurs informatiques. Cependant, le système nécessitait une adaptation pour chaque LABM et a montré plusieurs limites, sous-estimées lors de l’attribution du marché au prestataire informatique retenu. La principale était liée au format texte brut des données reçues à l’InVS. Le cahier des charges prévoyait la transmission de données codées accompagnées de dictionnaires, qui n’ont jamais pu être fournis. Le transcodage alors rendu nécessaire, long et fastidieux à mettre en œuvre, pouvait être invalidé par des variations minimes (ajout ou retrait d’un caractère) dans les libellés récupérés par le système. Les mêmes difficultés ont été observées pour le rapprochement des résultats de culture et des antibiogrammes. L'étude de faisabilité n’avait pas révélé ces difficultés, les données ayant été directement extraites des bases de données des LABM. La récupération des données à travers le flux continu d’impression constituait la seconde limite majeure, car la capture devait être instantanée, sans possibilité de rattrapage en cas de dysfonctionnement.
Les besoins en matière de transmission automatisée des données des laboratoires sont récurrents pour la surveillance épidémiologique et dépassent le cadre de la surveillance de la RATB. À l’InVS, deux expériences ont été conduites de façon concomitante au projet Labville : le système multi-sources cancer (SMSC) 13 et le réseau Oscour® de surveillance des passages aux urgences 14,15. Le SMSC était un projet pilote pour tester la mise en relation de trois sources de données : deux bases de données médico-économiques (programme de médicalisation des systèmes d’information (PMSI) et affections de longue durée (ALD) et une base de données des laboratoires d’anatomo-cytopathologie. Ce projet a rencontré des difficultés similaires : il a souligné l’absence de normalisation et de structuration des données des laboratoires d’anatomo-cytopathologie, les restrictions d’accès aux données gérées par des logiciels spécialisés et la difficulté de conduire un marché impliquant une multitude de partenaires. À court terme, ces travaux se poursuivront par le test de la mise en relation des deux bases médico-économiques. A contrario, Oscour® est le seul système qui produit aujourd’hui des données en routine de recours aux services d’urgence. Initié dans un contexte de crise, après la canicule de 2003, il a d’abord inclus des centres sans objectif de représentativité, ce qui a permis de valider progressivement ses éléments techniques. L’InVS a financé la mise en œuvre de requêtes d’extraction spécifiques de chaque éditeur de logiciel pour les services participants, et les données d’activité des services d’urgence, plus simples car agrégées, étaient transmises une fois par jour, en couvrant une fenêtre de sept jours pour pallier à d’éventuels défauts de transmission. Une mise en œuvre pragmatique et un volume restreint de données extractibles de bases de données structurées ont été des éléments clefs pour la réussite de ce projet.
En conclusion, l’expérimentation Labville a confirmé que les LABM étaient une source de données pertinente pour surveiller la RATB en ville mais n’a pas validé de solution technique pérenne pour leur exploitation en routine. Normalisation des bases de données, réforme de la biologie et accréditation des LABM pourront apporter des solutions à moyen ou long terme. Cependant, des approches pragmatiques sont indispensables pour répondre aux enjeux à court terme de la surveillance de la résistance aux antibiotiques en ville, notamment pour orienter les mesures de maîtrise de la diffusion croissante de souches d’E. coli productrices de bêtalactamases à spectre étendu (BLSE). Elles incluent la conduite d’études ponctuelles ciblées, éventuellement répétées, et la production d’indicateurs propres à la ville en collaboration avec les réseaux existants de laboratoires ou de médecins généralistes et les Centres nationaux de référence.
Remerciements
L’équipe Labville remercie
- l’ensemble des biologistes listés ci-après pour leur participation au réseau Labville :
Dr Megevand (Bourg-en-Bresse), Dr Pokorny (Saint-Quentin), Dr Guerin (Le Cannet), Dr Piquemal (Troyes), Dr Grellet (Aix-en-Provence), Mme Heboyan (Aix-en-Provence), Dr Fesquet (Marseille), Dr Porsin Weber (Bourges), Drs Jegouzo - Camus (Ussel), Dr Vialle (Bastia), Dr Moulinier (Besançon), Dr Becam (Quimper), Dr Segeral (Chateaulin), Dr Corre (Chateaulin), Dr Galinier (Toulouse), Dr Laurent (Cenon), Dr Hernandez (Lamalou-les-Bains), Dr Lagarde (Perols), Dr Baisset (Rennes), Dr Cahiez (Chateauroux), Dr Cadoux (Grenoble), Dr Perard (Grenoble), Dr Ciapa (Echirolles), Dr Touzet (Dole), Dr Hache (Roanne), Dr Mahfoudi (Brives-Charensac), Dr Langeard (Chateaubriant), Dr Dehorne (La Chapelle sur Erdre), Dr François Maillet (Saint-Herblain), Dr Graveron (Fleury-les-Aubrais), Dr Fons (Mende), Dr Delage (Carentan), Dr Roblin (Coutances), Dr Barrois (Epernay), Dr Tang (Vitry-le-François), Dr Bourdette (Maxeville), Dr Dumur (Bar-le-Duc), Dr Bize (Thionville), Mr Sukno (Lille ), Dr Naepels (Dunkerque), Dr Lionne (Roubaix), Dr Mathieu (Lens), Dr Chatron (Clermont-Ferrand), Dr Uthurriague (Pau), Dr Audry (Tarbes), Dr Klein (Obernai), Dr Nowak (Wissenbourg), Dr Pasteau (Mulhouse Kingersheim), Dr Pirat (Condrieu), Dr Coquard (Autun), Dr Perrut (Autun), Dr Mahe (Le Mans), Dr Berlioux (Paris), Dr Napoly (Paris), Dr Jaquot-Denis (Paris), Dr Moreau (Paris), Dr Vigier (Paris), Dr Szlafmyc (Paris), Dr Thibaud (Le Havre), Drs Rigal - Lamy (Rouen), Dr Bonhoure (Nemours), Dr Crouzier (Tournan-en-Brie), Dr Marque (Mantes-la-Jolie), Dr Benech (Caussade), Dr Laverdure (Montauban), Dr Schlegel (Draguignan), Dr Pescheux (Toulon), Dr Bandelier (Toulon), Dr Lhomme (Poitiers), Dr Grau (Poitiers), Dr Hangard (Saint Léonard de Noblat), Dr Grillet (Tonnerre), Dr Savie (Sens), Dr Guelpa (Yerres) (liste actualisée au 10 novembre 2009, classement par ordre de numéro de département). - les membres du comité de suivi Labville listés ci-après pour leurs conseils experts bienveillants :
Dr Philippe Chatron, Pr Patrice Courvalin, Dr Jean-Claude Desenclos, Dr Emmanuelle Espié, Dr Gilles Fesquet, Dr Fréderic Laurent, Dr Anne Gallay, Dr Frédéric Grobost, Dr Agnès Lepoutre, Dr Françoise Moreau, Dr Emmanuelle Varon. - le Dr Hélène Aubry-Damon pour son initiation du projet Labville et Maëlaïg Mevel pour son suivi des premiers équipements de LABM.
Ce travail a été financé par l’Institut de veille sanitaire, sans recours à des financements extérieurs.