Épidémiologie de l’infection à Helicobacter pylori en France en 2020 : données de surveillance du CNR Campylobacters et Hélicobacters
// Epidemiology of Helicobacter pylori infection in France in 2020: Surveillance data from the National Reference Centre for Campylobacters and Helicobacters
Résumé
Le Centre national de référence des Campylobacters et des Hélicobacters (CNRCH) travaille avec un réseau de correspondants locaux et nationaux. L’activité H. pylori au CNRCH a poursuivi sa progression en 2020 grâce en particulier au recrutement de nouveaux correspondants. L’année 2020 confirme la stabilisation de la résistance primaire aux macrolides et aux fluoroquinolones. De rares souches résistantes à l’amoxicilline ou à la rifampicine ont été identifiées en 2020. Nos données démontrent l’intérêt d’associer la recherche par culture de cette infection à une détection moléculaire de la bactérie et des mutations associées à la résistance aux macrolides. Notre participation à un protocole de surveillance français et à un réseau de surveillance européen nous permet de comparer nos données internes au CNR avec des données obtenues sur le territoire national et européen. L’année 2020 a été marquée par une forte adhésion de nos correspondants à nous fournir des renseignements cliniques associés à l’envoi de biopsies gastriques.
Abstract
The National Reference Center for Campylobacters and Helicobacters (NRCCH) works with a network of local and national correspondents. The H. pylori activity at the NRCCH continued to increase in 2020 thanks to the recruitment of new correspondents. The year 2020 confirms the stabilization of primary resistance to macrolides and fluoroquinolones. Rare strains resistant to amoxicillin and rifampicin were identified in 2020. Our data demonstrate the value of combining culture-based investigation of this infection with molecular detection of the bacteria and the mutations associated with macrolide resistance. Our participation in a French and a European surveillance network allows us to compare our internal data at the NRCCH with data obtained throughout the entire French territory and abroad. The year 2020 was also marked by strong support from our correspondents in providing us with clinical information associated with the submission of gastric biopsies.
Introduction
Helicobacter pylori a été cultivé pour la première fois en 1982. L’intérêt pour cette bactérie a augmenté progressivement durant la décennie 80 pour devenir très important dans la décennie suivante quand son rôle dans les maladies gastroduodénales a été démontré. Il est maintenant avéré que l’infection à H. pylori est la cause essentielle de la maladie ulcéreuse. De plus, il s’agit de la première infection bactérienne à l’origine de cancers chez l’homme : 80% des lymphomes gastriques du MALT (mucosa-associated lymphoid tissue) sont dus à H. pylori et peuvent être guéris par éradication de cette bactérie (sauf aux stades trop avancés avec translocations associées). Concernant les carcinomes gastriques distaux, l’infection à H. pylori constitue un facteur de risque majeur présent dans plus de 90% des cas.
Depuis les années 90, des traitements efficaces se sont développés et des conférences de consensus se sont tenues sur tous les continents pour recommander quand et comment éradiquer cette infection.
La surveillance épidémiologique des infections à H. pylori fait partie des missions du Centre national de référence des Campylobacters-Hélicobacters (CNRCH). Le CNRCH publie régulièrement des études permettant de caractériser les performances des différentes techniques de diagnostic de cette infection et de suivre l’évolution des résistances aux antibiotiques. L’activité du CNRCH a augmenté de manière significative depuis 2017, en phase avec la parution de recommandations de la Haute Autorité de santé (HAS) concernant la prise en charge du diagnostic de cette infection (1). Le réseau de surveillance du CNRCH permet d’avoir une vision nationale de l’épidémiologie de l’infection à H. pylori et des résistances primaires et secondaires aux antibiotiques. Les données présentées dans cet article concernent l’année 2020.
Matériels et méthodes
Description du réseau H. pylori
Le CNRCH reçoit des biopsies qui lui sont adressées en première intention pour un diagnostic bactériologique de l’infection à H. pylori. Le CNRCH réalise en systématique une culture et une détection moléculaire de la bactérie et des mutations associées à la résistance aux macrolides. Les recommandations de l’HAS de 2017 n’ont pas modifié ces pratiques.
Le CNRCH reçoit des biopsies gastriques en provenance du centre hospitalier universitaire (CHU) de Bordeaux et de correspondants externes au CHU de Bordeaux répartis dans 32 hôpitaux et cliniques français, soit 30 départements en 2020 et 1 DOM (La Réunion). L’activité H. pylori du CNRCH est liée à celle de ses correspondants externes représentant plus de 90% des biopsies reçues (95,1% en 2020, 92% en 2019). Le réseau est entretenu et développé grâce aux efforts conjoints des biologistes, des techniciens et du personnel administratif du CNRCH. Courant 2020, le CNRCH a intégré 21 nouveaux correspondants dans le réseau : 5 CHU et 16 centres hospitaliers généraux (CHG). Le réseau fonctionne sur la base du volontariat, sans obligation d’envoi systématique des biopsies gastriques au CNRCH. Le CNRCH reçoit donc des biopsies de laboratoires des cinq régions françaises qui ne souhaitent pas ou ne peuvent pas techniquement assurer le diagnostic bactériologique de l’infection à H. pylori. La répartition par région est hétérogène avec trois régions représentant 96,2% des biopsies reçues (Nouvelle-Aquitaine 79%, Grand Est 12,5% et Île-de-France 4,7%).
Culture de H. pylori à partir de biopsies gastriques
Au cours de l’endoscopie gastrique, plusieurs biopsies sont prélevées dans l’antre à environ 3 cm du pylore et au niveau du tiers supérieur du fundus. Les échantillons doivent être adressés rapidement en utilisant un milieu de transport spécifique à +4°C (milieu Portagerm Pylori, bioMérieux) : le délai de transport doit être idéalement de 24h 1. À leur arrivée, les biopsies sont broyées avec du matériel jetable (microtube + pilon) dans un bouillon nutritif.
Afin de suivre les pathologies associées à ces infections, le CNRCH a mis en place courant 2018 une fiche de renseignements devant accompagner les biopsies gastriques envoyées au CNRCH (2). Les données collectées par l’analyse des fiches de renseignements cliniques nous permettent d’estimer la résistance primaire aux antibiotiques (patients naïfs de traitement d’éradication) et secondaires (patients en échec d’au moins un traitement d’éradication).
Le produit de broyage est ensuite ensemencé sur un milieu constitué d’une base gélosée (milieux cœur-cervelle, Columbia, Wilkins-Chalgren ou Brucella, par exemple) additionnée de 10% de sang (humain ou à défaut mouton ou cheval). Des suppléments sélectifs sont utilisés pour inhiber la croissance de contaminants occasionnels. L’incubation est réalisée rapidement en atmosphère microaérobie, humide, à 37°C. En primoculture, les colonies n’apparaissent généralement pas avant trois jours. Les primocultures doivent être incubées 10-12 jours avant d’être déclarées négatives.
L’identification à l’espèce est facile sur les critères d’exigence culturale (microaérobie), de par l’aspect incurvé ou spiralé au Gram et de la présence d’une activité uréasique, oxydasique et catalasique.
Les antibiogrammes sont réalisés par la détermination des concentrations minimales inhibitrices (CMI) par méthode du E-test® selon les recommandations du CASFM (3) (CA-SFM/EUCAST 2021) :
–milieu MH-10% sang de mouton 1 ;
–inoculum : 3 McFarland ;
–incubation : atmosphère microaérobie, 37°C, 48h. Si la culture est insuffisante après 48h, réincuber immédiatement et effectuer une lecture après 72h d’incubation.
Un contrôle de qualité est effectué à chaque changement de lot de gélose avec la souche Helicobacter pylori CCUG 17874. Le CNRCH participe à un échange annuel inter-laboratoires avec les CNR européens concernant les antibiogrammes de H. pylori afin de vérifier la conformité de ses résultats. (4)
Détection par PCR de H. pylori et des mutations associées à la résistance aux macrolides
Le CNRCH utilise une PCR maison développée sous format FRET 2 qui a été adaptée courant 2019 en barrettes prêtes à l’emploi synthétisées par la société Eurogentec (Liège, Belgique). Le CNRCH participe au Quality Control for Molecular Diagnosis (QCMD) pour la PCR H. pylori afin de vérifier la conformité de ses résultats.
Surveillance des mutations présentes dans le gène rpoB associées à la résistance aux rifamycines chez H. pylori
Le gène rpoB de souches H. pylori ayant une CMI à la rifampicine supérieure à 4 mg/L est séquencé systématiquement afin de déterminer les mutations responsables de cette résistance ; 330 paires de bases du gène rpoB sont amplifiées et séquencées à l’aide des amorces FrpoB (TTTGATTCGCTCATGCCCCAT) et RrpoB (CACAACCTTTTTATAAGGGGC) 3.
Évaluation des mutations associées à la résistance à l’amoxicilline chez H. pylori
Les souches de H. pylori présentant une CMI à l’amoxicilline supérieure à 0,125 mg/L sont catégorisées résistantes. Le gène pbp1, codant pour la « penicillin-binding protein », est connu pour être responsable de la résistance à l’amoxicilline, mais les mutations en cause sont nombreuses 4. Afin de qualifier les souches présentant une CMI augmentée à l’amoxicilline, l’intégralité du gène pbp1 de ces souches est amplifiée avec les amorces F1pbp1 (CGGTTTCTAAACCCCTTTTACG) et R1pbp1 (AGCGGTGAGTATCCTTGTGG), et séquencée avec F1pbp1, R1pbp1 et les quatre amorces internes F2pbp1 (ACCTTGCAAGAAGCCTTGAG), R2pbp1 (AACTTGGAGCCCTAGGCAAG), F3pbp1 (AAACATGGTGCTCACACGAG), R3pbp1 (TTCAAAGCCAAGCTGATCG).
Résultats
Description du réseau de partenaires et des prélèvements reçus
En 2020, le CNRCH a reçu 1 885 biopsies gastriques pour recherche par culture et PCR de H. pylori. Ainsi, en 2020, l’activité concernant la recherche de H. pylori par culture et PCR n’a pas chuté malgré la période de confinement liée à la crise du Covid-19, avec une augmentation de 4,6% du nombre de biopsies reçues. Pour quelques rares cas, la culture ou la PCR ont été réalisées isolément : réception de souche déjà identifiée (22 soit 0,9%) ou d’ADN déjà extrait (8 soit 0,3%). Si l’on se base sur les résultats de la culture et/ou de la PCR, 32,4% des prélèvements reçus étaient positifs en 2020 pour H. pylori.
Caractéristiques de la population de patients
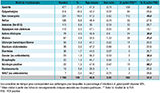
Pour 175 patients, 2 prélèvements (antre et fundus) ont été reçus, soit 1 885 biopsies reçues pour 1 740 patients. Pour ces 1 740 patients, l’âge moyen était de 46,9 ans avec un sex-ratio H/F moyen de 0,65. La moyenne d’âge était relativement homogène en fonction du motif d’endoscopie (excepté les populations consultant pour anémie, ulcère, cancer ou MALT dont les moyennes d’âge dépassent 50 ans), tout comme le sex-ratio (excepté pour les ulcères et les patients consultant pour vomissements avec un sex-ratio >1) (tableau 1). Selon les données obtenues à partir des biopsies gastriques, la majorité des cas positifs (68,7%) avaient entre 30 et 69 ans et étaient majoritairement des femmes dans quasiment toutes les tranches d’âge (excepté pour la tranche des 40-49 ans où le sex-ratio H/F moyen était à 0,82).
Nous disposons pour 2020 des renseignements cliniques associés à ces 1 740 patients (tableau 1). La majorité des patients consultaient pour des gastrites ou des épigastralgies. Ceux présentant des ulcères et les patients orientés vers une endoscopie digestive haute à cause d’une sérologie H. pylori positive, étaient les plus fréquemment infectés.
 Agrandir l'image
Agrandir l'imageSurveillance de la résistance de H. pylori aux anti-infectieux
En 2020, sur les 443 souches de H. pylori détectées par culture, 15 n’ont pas pu être testées à cause d’une subculture négative.
La résistance à l’amoxicilline, rifampicine et tétracycline est rare ou nulle chez H. pylori. Le tableau 2 récapitule les principaux phénotypes de résistance retrouvés en 2020 : près d’un tiers des souches étaient sensibles à tous les antibiotiques testés et 35% environ résistantes isolément au métronidazole. Les souches isolément résistantes à la clarithromycine ou à la lévofloxacine ne représentaient que respectivement 4,7% et 3,5%. Toutes les souches résistantes à la rifampicine ou à l’amoxicilline cumulaient 2 à 3 marqueurs de résistance supplémentaires.
 Agrandir l'image
Agrandir l'imageLes données présentées dans le tableau 3 comparent les résultats obtenus par E-test® et ceux obtenus par PCR de détection des mutations associées à la résistance aux macrolides et ce pour 427 des 428 souches viables.
 Agrandir l'image
Agrandir l'imageLes discordances sont rares entre phénotype et génotype : 0,5% uniquement de discordance majeure due à deux cas interprétés Résistant in vitro mais de génotype « Wild Type » (WT, sauvage) par PCR. Pour 8 cas, seule la population sensible a été retrouvée sur l’antibiogramme alors que la PCR a détecté la présence d’une double population A2142-43G + WT. Ceci pourrait être interprété comme une discordance mineure (1,9%). Dans 33 cas, c’est la population résistante qui a été vue par culture alors que la PCR détectait une double population A2142-43G + WT.
L’ensemble de ces résultats démontre l’intérêt en routine de coupler culture (avec antibiogramme) et PCR.
Selon les résultats de PCR (tableau 4), le génotype A2142-43G est le plus fréquent et la proportion de double population (WT+R) modérée. La mutation A2142C reste anecdotique en France. La mutation A2142T décrite en 2018 5, n’a pas été retrouvée depuis.
 Agrandir l'image
Agrandir l'imageLe gène rpoB de quatre souches de H. pylori résistantes à la rifampicine a été séquencé afin de déterminer les mutations responsables de cette résistance : la mutation D530N a été retrouvée trois fois, la quatrième souche présentait une mutation H540Y (CMI>32 mg/L pour ces quatre souches).
Pour les 1 740 patients, nous avons recueilli pour 1 440 d’entre eux une information concernant un traitement d’éradication antérieur : 1 344 étaient naïfs de tout traitement d’éradication (93,3%) et 196 avaient reçu au préalable une ou plusieurs lignes de traitement d’éradication (13,6%). La PCR H. pylori était positive pour 484 patients (392 patients naïfs et 92 patients en échec d’éradication) : ces données nous ont permis d’estimer les pourcentages de résistance primaire et secondaire à la clarithromycine (tableau 5). La culture, couplée avec un antibiogramme complet pour tous les antibiotiques testés, était positive pour 368 patients (291 patients naïfs et 77 patients en échec d’éradication) : ces données nous ont permis d’estimer les pourcentages de résistance primaire et secondaire aux autres antibiotiques d’intérêt pour le traitement d’éradication de l’infection à H. pylori.
Le pourcentage de résistance primaire aux macrolides était de 19,9% versus 47,8% en secondaire (tableau 5) et pour la lévofloxacine 17,1% de résistance primaire versus 15,6% en secondaire.
 Agrandir l'image
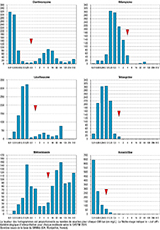
Agrandir l'imageDepuis 2016, le CNR surveille la distribution des CMI pour toutes ces molécules. Les données présentées ci-après montrent la répartition des CMI pour 1 555 souches de H. pylori et intègrent celles de 2020 (figure). Les « cut-offs » épidémiologiques utilisés pour interpréter les antibiogrammes de H. pylori semblent toujours adaptés. Selon les recommandations du Groupe d’étude français des Hélicobacters (GEFH) (5), du CNRCH et du CASFM, les antibiotiques qui doivent être testés (en CMI uniquement) sont la clarithromycine et la lévofloxacine. Il n’est pas nécessaire de tester l’amoxicilline car cette résistance est rare. La sensibilité à la tétracycline et aux rifamycines peut être testée en seconde intention bien que les résistances soient rares.
Même si le CASFM ne recommande pas de tester en routine la sensibilité au métronidazole, du fait du manque de reproductibilité des résultats de CMI inter-laboratoires et de sa signification clinique limitée, le « cut-off » historiquement utilisé (8 mg/L) semble bien séparer les deux populations de souches distinctes. Toute résistance à la tétracycline doit être vérifiée. Plus de 60% des souches présentent une CMI <0,016 mg/l à l’amoxicilline : toute résistance doit être également contrôlée (figure).
 Agrandir l'image
Agrandir l'imageDiscussion-Conclusion
L’infection à H. pylori en France est prise en charge en majorité par des généralistes et gastroentérologues. Les conférences de consensus, les recommandations de l’HAS et des sociétés savantes (Groupe d’études français des Hélicobacters, Société nationale française de gastroentérologie) sont des guides précieux dans leur pratique (6). Les données générées par le CNRCH apportent des éléments d’information complémentaires afin d’évaluer l’impact de ces recommandations et suivre l’évolution des résistances aux antibiotiques chez H. pylori.
La majorité des patients de 2020 souffraient de gastrite (27,4%) mais seuls 33,3% étaient infectés. En revanche 68,2% de patients pour lesquels la recherche a été motivée par une sérologie positive étaient toujours infectés par H. pylori. Ceci reflète bien l’application par les cliniciens des recommandations de l’HAS qui recommande la sérologie comme test diagnostic de première intention pour les patients sans symptôme digestif de moins de 40-45 ans, apparentés à un patient ayant eu un cancer gastrique ou avec antécédent d’ulcère sans preuve d’éradication de H. pylori (y compris avant prise d’AINS ou d’aspirine à faible dose), ou avec purpura thrombopénique immunologique. En cas de positivité, ces recommandations conseillent de pratiquer une endoscopie avec envoi de biopsies pour culture et PCR.
La gastroscopie avec envoi des biopsies pour examen histopathologique et culture (±PCR) est également recommandée en première intention pour les patients avec symptômes orientant vers une pathologie digestive haute notamment : syndrome ulcéreux, dyspepsie chez un patient >40-45 ans et/ou en cas de symptômes d’alarme (dont dysphagie, amaigrissement, anémie), anémie ferriprive ou carence en vitamine B12 sans cause trouvée, patients avec facteurs de risque de cancer gastrique (personnes >40-45 ans, apparentées à un patient ayant eu un cancer gastrique), lymphome gastrique du MALT, intervention bariatrique prévue.
Les données cliniques du CNRCH de l’année 2020 sont le reflet de l’application de ces recommandations.
En dehors des laboratoires possédant une bonne expertise dans le domaine du diagnostic des infections à H. pylori (CHU de Poitiers, et APHP-Cochin notamment) peu de laboratoires en France réalisent la recherche de cette infection à partir de biopsies gastriques. La principale raison tient à la difficulté de cultiver cette bactérie fragile et de maitriser en particulier des conditions pré-analytiques adaptées à la survie de cette bactérie. Le CNRCH met à disposition sur son site Internet des fiches pratiques conseils pour aider les microbiologistes à maîtriser cette culture (4). La PCR H. pylori n’est malheureusement toujours pas remboursée. Plusieurs kits de PCR temps réel commerciaux performants sont pourtant disponibles en France. Les données 2020 montrent comme attendu que la PCR est plus sensible que la culture pour la recherche de l’infection à H. pylori et que la corrélation phénotype/génotype pour la détermination de la sensibilité à la clarithromycine est excellente. La culture en association à la PCR permet dans de rares cas de détecter des doubles populations de souches présentant des profils de sensibilité mixte à la clarithromycine et de répondre pour la sensibilité aux autres molécules d’intérêt pour le traitement d’éradication notamment la lévofloxacine. Pour cette dernière, aucun kit de PCR temps réel ne permet la détection des mutations présentes dans la « Quinolone Resistance Determining Region » de gyrA.
La PCR pourrait être réalisée en primo diagnostic d’un patient naïf de tout traitement d’éradication. La culture serait réservée en cas de détection d’une mutation associée à la résistance à la clarithromycine. La culture devrait être réalisée en première intention en cas d’échec de traitement.
Les données 2020 éclairent sur l’évolution de la résistance aux antibiotiques de H. pylori en France. La résistance aux macrolides est proche de 20%. De par la distribution géographique hétérogène des biopsies reçues en 2020 au CNRCH, il était important de comparer nos résultats à ceux des précédentes études afin de vérifier la représentativité de nos données de surveillance, notamment pour la résistance aux antibiotiques. Les données sont similaires également à celles d’une étude menée par le CNRCH et publiée récemment : l’étude PHARE 6. Il s’agit d’une étude observationnelle menée en 2018 incluant des biopsies gastriques obtenues de 62 gastroentérologues sélectionnés au hasard dans cinq régions de France et envoyées au CNRCH. Tout comme pour notre réseau de surveillance 2020, les gastroentérologues des régions Nouvelle-Aquitaine, Grand Est et Île-de-France ont envoyé le plus de biopsies.
La résistance primaire à la clarithromycine était de 20,9%, à la lévofloxacine de 17,6% et au métronidazole de 58,6%. La résistance primaire à la clarithromycine qui était passée de 22,2 à 20,3% entre 2014 et 2016 semblait donc se stabiliser autour de 20%. Malgré l’utilisation de Pylera® qui est le seul traitement incluant cet antibiotique, aucune souche résistante à la tétracycline n’était détectée en France. Le pourcentage de résistance primaire à la lévofloxacine dépasse également 15%. Aussi ni la clarithromycine, ni la lévofloxacine ne peuvent être prescrites en probabiliste.
Aucune augmentation entre résistance primaire à la lévofloxacine versus résistance secondaire n’a été observée en 2020. Ceci est probablement dû au fait que la lévofloxacine n’est pas conseillée dans les traitements probabilistes. La résistance primaire et secondaire au métronidazole est élevée en France.
Le Groupe européen Helicobacter et Microbiote (EHMSG) a également publié cette année une étude menée en 2018 qui avait pour but d’apprécier l’évolution de la résistance aux antibiotiques de H. pylori dans la plupart des pays européens 7. Les taux de résistance pour les 1 211 patients adultes inclus étaient de 21,4% pour la clarithromycine, 15,8% pour la lévofloxacine et 38,9% pour le métronidazole. Une association significative a été trouvée entre la résistance à la clarithromycine chez H. pylori et la consommation dans la communauté des macrolides et entre la résistance à la lévofloxacine et la consommation de quinolones. Ce même type d’étude serait pertinent pour analyser les données 2020 d’antibiorésistance de H. pylori en France.
Les forts taux de résistances primaires à la clarithromycine et à la lévofloxacine ne permettent pas l’utilisation de ces molécules sans test de sensibilité préalable pour le traitement de H. pylori. L’impact de la résistance au métronidazole est plus limité, surtout si on allonge la durée de traitement. Le CNRCH conjointement avec le GEFH milite pour une stratégie d’éradication orientée soit par PCR soit basée sur le résultat d’un antibiogramme. Nous constatons d’ailleurs en 2020 que les souches résistantes isolément à la clarithromycine ou à la lévofloxacine sont rares par comparaison aux souches cumulant plusieurs marqueurs de résistance. Les mutations retrouvées associées en 2020 à la résistance à la rifabutine sont connues et décrites 8. Celles dans pbp1 associées à la résistance à l’amoxicilline sont en cours d’étude en collaboration avec l’Institut Pasteur (Paris).
En conclusion, l’application des recommandations de recherche et de traitement de l’infection à H. pylori semble s’harmoniser en France. Le CNRCH continue ces efforts pour contribuer à la formation des biologistes, à valider de nouvelles techniques, et à conseiller les cliniciens.
Remerciements
Le CNRCH remercie tous les techniciens du laboratoire de Bactériologie du CHU de Bordeaux, Hôpital Pellegrin qui participent à la recherche par culture de H. pylori dans les biopsies gastriques. Ces données présentées dans cet article existent grâce aux correspondants du CNRCH qui nous font confiance en nous envoyant leurs biopsies gastriques accompagnées de renseignements cliniques. Votre confiance en notre expertise nous honore. Le secrétariat du CNRCH assure un lien essentiel entre notre centre et nos correspondants.
Liens d’intérêt
Les auteurs déclarent ne pas avoir de liens d’intérêt au regard du contenu de l’article.