Twitter : un outil complémentaire pour la surveillance de l’épidémie saisonnière de grippe en France métropolitaine et en région ?
// Twitter: a complementary tool for monitoring the seasonal influenza epidemic in metropolitan France and the regions?
Résumé
Introduction –
Aujourd’hui, les médias sociaux comme Twitter sont utilisés par les individus pour diffuser de l’information en santé mais également pour partager ou échanger sur leur état de santé. Dans ce contexte, une étude exploratoire a été mise en place afin d’étudier si les données de Twitter peuvent être utilisées comme un proxy pour la surveillance de l’épidémie saisonnière de grippe en France, et plus particulièrement à l’échelle régionale.
Méthodes –
Un système automatisé permettant le recueil en temps réel et un prétraitement (géolocalisation et classification) de tweets relatifs à la grippe a été développé. Les données recueillies sur la période du 8 août 2016 au 26 mars 2017 ont ensuite été comparées à celles du système de surveillance syndromique SurSaUD® (réseaux OSCOUR® et SOS Médecins).
Résultats –
Au total, sur la période d’étude, le système a permis de recueillir 238 244 tweets relatifs à la grippe, dont 130 559 étaient localisés en France. Après une étape de nettoyage, l’algorithme a permis de classer 22 939 tweets indiquant un état grippal. Une corrélation positive et statistiquement significative a été observé entre les tweets indiquant un état grippal et les consultations pour grippe/syndrome grippal rapportées par les réseaux OSCOUR® et SOS Médecins, à la fois au niveau national et au niveau régional.
Discussion – conclusion –
Cette étude exploratoire a permis de montrer que les données de Twitter, en complément des systèmes existants, permettent le suivi de l’épidémie saisonnière de grippe en France et en région. Le système devra être amélioré pour confirmer les tendances observées lors de la prochaine épidémie de grippe.
Abstract
Introduction –
Social media as Twitter are used today by people to disseminate health information, but also to share or exchange on their health condition. In this context, an exploratory study was implemented to investigate whether Twitter data can be used as a proxy for monitoring the seasonal influenza epidemic in France and at the regional level.
Methods –
A real-time automated system allowing the collection, the pre-processing (geolocation and classification) of tweets related to influenza illness was developed. The data collected from 8 August 2016 to 26 March 2017 were compared to those of the French syndromic surveillance system SurSaUD® (OSCOUR® and SOS Médecins networks).
Results –
Over the study period, the system collected 238,244 influenza-related tweets of which 130,559 were located in France. After a cleaning step, 22,939 tweets were classified by the algorithm as an influenza-like illness. A positive and statistically significant correlation between tweets and ILI consultations reported by the OSCOUR® and SOS Médecins networks was identified at both national and regional levels.
Discussion – conclusion –
This exploratory study allowed to show that Twitter data can be used to monitor the epidemic of seasonal influenza in France and at the regional level, in complementarity with existing systems. The system needs to be improved to confirm the trends observed during the next influenza epidemic.
Introduction
Au cours de ces dernières années, l’évolution des technologies numériques, et en particulier du réseau Internet, a bouleversé notre manière de communiquer, d’accéder à l’information et de la partager. Aujourd’hui, le « Web 2.0 » permet aux internautes d’interagir ensemble, de créer du contenu Web, de l’organiser, de le modifier et de le combiner avec des créations personnelles (par opposition au Web 1.0 où l’information était unidirectionnelle, plaçant l’internaute dans une attitude passive). Depuis quelques années, le terme Web 2.0 tend à être remplacé par l’expression « médias sociaux » qui regroupe les nouvelles technologies sociales tels que les réseaux sociaux de contacts professionnels ou généralistes, les forums, les blogs, les wikis, les réseaux sociaux de contenu (microblogs, partage de vidéos, des photos, etc.). Désormais, les médias sociaux sont utilisés par les individus pour communiquer, maintenir des liens avec leur famille et leurs amis, discuter, diffuser de l’information et apprendre.
L’essor des médias sociaux n’a pas échappé au domaine de la santé : les patients et leur entourage peuvent échanger directement, confronter leurs points de vue, rechercher de l’information médicale, décrire leurs expériences et leurs symptômes sans passer par les médecins, experts, scientifiques, institutions. Les médias sociaux constituent ainsi pour les épidémiologistes une source informelle complémentaire de données, qui peuvent être utilisées pour identifier des informations de santé non recueillies ou en complément des systèmes de surveillance traditionnels et révéler les points de vue sur des sujets liés à la santé. De ce constat, une nouvelle approche scientifique est développée : l’infodémiologie. L’infodémiologie peut être définie comme la science basée sur des technologies de l’information et des communications dont l’objectif est de surveiller l’état de santé des populations et d’orienter les politiques de santé publique 1.
Parmi les services de réseautage en ligne, Twitter est une plateforme gratuite de microbloging permettant aux utilisateurs de diffuser des messages courts appelés « tweets » et d’échanger des informations, des photos ou vidéos et des liens Internet. Chaque tweet publié est limité à 280 caractères depuis septembre 2017 (140 caractères auparavant) 2. Par défaut, les tweets sont publics, néanmoins l’utilisateur a la possibilité d’écrire des messages privés à des followers sélectionnés. Des études récentes ont montré que les données de Twitter pouvaient être employées pour la surveillance de pathologies infectieuses comme la grippe 3,4,5,6,7, le choléra 8 ou la dengue 9,10.
Dans ce contexte, une étude exploratoire a été mise en place afin d’étudier si les données de Twitter pourraient être utilisées comme un proxy pour la surveillance de l’épidémie saisonnière de grippe en France, et plus particulièrement à l’échelle régionale.
Matériel et méthodes
Twitter met à disposition des développeurs différents protocoles de communications ou API (interfaces de programmation) qui permettent d’interroger sa base de données mais également d’utiliser certaines fonctionnalités. Pour cette étude, nous avons utilisé l’API Streaming, qui permet d’extraire en temps réel un ensemble de messages à partir de mots-clés. Cette API renvoie un fichier JSON (JavaScript Object Notation) qui peut être exploité par différents logiciels, dont le logiciel R. Le fichier des données contient des informations sur les tweets (date de publication, texte du message, lien Internet, etc.), le compte de l’utilisateur (date de création du compte, pseudo…) et des informations de localisation (longitude/latitude). L’accès gratuit et anonyme à cette source de données limite le volume de tweets reçus à 1% du volume total de tweets publiés. Autrement dit, les requêtes couvrant moins de 1% du volume de tweets publiés à chaque instant sont censées renvoyer l’intégralité des tweets correspondants. Au-delà, un échantillonnage non-aléatoire est réalisé par Twitter 11.
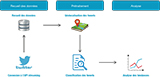
Un programme R permettant le recueil, le prétraitement et l’analyse des tweets a été développé (figure 1).
Recueil des données
La première étape consistait à se connecter sur l’API Streaming de Twitter et, au moyen d’une requête, à recueillir : les tweets en langue française contenant les mots-clés « grippe », « grippaux », « grippal », sans préciser une géolocalisation en particulier. En effet, une étude a montré que les tweets géolocalisés ne représentaient qu’environ 2% de tous les tweets et 3% des utilisateurs de Twitter 12. Afin de ne retenir que les tweets relatifs à la forme humaine de la grippe, tous les tweets contenant les mots suivants ont été exclus de la base de données : « volaille », « aviaire », « volailles », « porc », « porcin ».
La période de recueil s’est étendue de la semaine 36 de 2016 (8-14 août) à la semaine 12 de 2017 (20-26 mars) afin de contenir à la fois un bruit de fond préalable et la totalité de la saison grippale en France métropolitaine.
Prétraitement
Géolocalisation des tweets
La communauté francophone qui s’exprime sur Twitter s’étend en dehors du territoire français. Un filtrage géographique des données était donc nécessaire. Pour identifier les tweets localisés en France, une combinaison de filtres automatisés a été implémentée 7. Dans un premier temps, tous les tweets comportant les coordonnées géographiques (coordonnées GPS, code du pays, le pays, la ville) de l’utilisateur lors de la publication du tweet ont été extraits. Lorsque les coordonnées de position n’étaient pas renseignées, d’autres données d’emplacement contenues dans le tweet (lieu indiqué dans le profil de l’utilisateur, fuseau horaire) ont été utilisées après normalisation de la chaine de caractères (suppression de certaines ponctuations, symboles, espaces supplémentaires, conversion de tous les mots en minuscules). Au final, ont été retenus :
–les tweets comportant des coordonnées géographiques localisées en France (coordonnées GPS, code du pays, le pays, la ville) ;
–les tweets dont le lieu indiqué dans le profil de l’utilisateur se rapportait à une ville, département ou région de France ;
–les tweets incluant le fuseau horaire de Paris, en excluant les tweets dont le lieu indiqué dans le profil utilisateur se situait hors de France.
À l’aide d’une table de correspondance, les tweets ont été automatiquement classés par région de France.
Classification des tweets relatifs à la grippe
Twitter peut être utilisé aussi bien par les individus pour s’exprimer sur leur état de santé que par des entreprises ou institutions pour diffuser ou relayer de l’information en santé. Dans cette étude, le défi majeur consistait donc à identifier, dans un gros volume de tweets, ceux relatifs à l’expression clinique de la grippe. Dans ce contexte, les tweets ont été filtrées à l’aide d’une procédure de classification automatique. Au préalable, un nettoyage du texte a été appliqué à chaque tweet : suppression des Retweets, des liens URL, des mots-dièses (« hashtags »), des accents, caractères spéciaux, de la ponctuation, et conversion de tous les mots en minuscules. Si un tweet ne contenait plus de texte après nettoyage, il était supprimé.
Pour filtrer les tweets, un classificateur de machine à vecteurs de support (SVM) a été utilisé. Pour former le classificateur, un échantillon aléatoire de 1 500 tweets a été extrait de la base de données. Chacun de ces tweets a été inspecté manuellement et étiqueté comme valide ou invalide selon la probabilité qu’il indiquait ou non un état grippal (tableau 1) 13.
Cet ensemble d’apprentissage codé manuellement a été converti en représentation vectorielle sous forme de fréquence inverse de document (TF-IDF) qui donne un poids plus important aux termes les moins fréquents, considérés comme plus discriminants. Ces vecteurs TF-IDF ont ensuite été introduits dans le SVM pour l’entraînement. Pour évaluer les performances du classificateur, le rappel, la précision et la F-mesure ont été calculés sur un échantillon aléatoire de 1 000 tweets (tableau 2).
 Agrandir l'image
Agrandir l'imageÀ partir du tableau 2, la précision a été calculée selon la formule suivante :
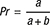
et le rappel était :
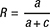
La F-mesure étant la moyenne harmonique de la précision et du rappel.
Analyse
Les données de Twitter ont été comparées à celles du dispositif SurSaUD® qui repose notamment sur deux sources de données, les structures d’urgence hospitalières participant au réseau OSCOUR® (Organisation de la surveillance coordonnée des urgences) et les associations SOS Médecins 14. Afin de tester l’existence d’une liaison entre le nombre hebdomadaire de tweets relatifs à la grippe et le nombre hebdomadaire de passages ou de consultations pour grippe, des coefficients de corrélation de Spearman et les tests de significativité ont été calculés pour chaque région de France.
Aspects éthiques
Conformément aux préconisations de la Commission nationale de l’informatique et des libertés (Cnil), toutes les informations sur le compte Twitter des utilisateurs ont été supprimées lors de la constitution de la base de données, excepté pour la variable localisation du compte, afin d’anonymiser les données personnelles des utilisateurs. Les noms d’utilisateurs pouvant apparaître dans le texte du tweet ont également été supprimés.
Résultats
Au total, sur la période du 8 août 2016 au 26 mars 2017, 238 244 tweets contenant les mots-clés « grippe », « grippal » « grippaux » ont été recueillis par le programme. L’analyse géographique a permis de localiser 130 559 tweets (54,8%) en France. Parmi eux, 3,4%(n=4 442) contenaient des coordonnées GPS, 83,0% (n=108 279) ont été localisés à partir du lieu indiqué dans le profil de l’utilisateur et 13,6% (n=17 838) à partir du fuseau horaire.
Après l’étape de nettoyage, 63 406 tweets ont été analysés par le classificateur sur la période d’étude. L’algorithme de classification testé sur un échantillon aléatoire de 1 000 tweets indiquait une F-mesure de 0,732, avec une précision de 0,739 et un rappel de 0,725. L’algorithme a classé 22 939 tweets comme valides, c’est-à-dire indiquant un état grippal.
Au total, la durée du processus de prétraitement (incluant la lecture du fichier JSON, de plus de 1 gigaoctet, par R) et de l’analyse des données (hors catégorisation manuelle des tweets) de la période d’étude a été évaluée à moins d’une heure.
La figure 2 montre que le nombre hebdomadaire de tweets relatifs à la grippe suit la même tendance que le nombre hebdomadaire de passages aux urgences et de consultations SOS Médecins pour grippe, à la fois au niveau national et au niveau régional, avec une augmentation parfois plus précoce pour les données de Twitter.
 Agrandir l'image
Agrandir l'imageQuelle que soit la source de données, les coefficients de corrélation de Spearman étaient positifs et statistiquement significatifs au niveau national et pour chaque région de France (tableau 3).
 Agrandir l'image
Agrandir l'imageAu niveau national, le coefficient de corrélation entre le nombre de tweets relatifs à la grippe et le nombre de passages aux urgences pour grippe était de 0,89 (p<0,001) et variait de 0,67 (p<0,001) pour la région Corse à 0,90 (p<0,001) pour la région Pays de la Loire. Le coefficient de corrélation était légèrement plus élevé pour SOS Médecins avec rs=0,90 (p<0,001) et il variait de 0,70 (p<0,001) pour la région Corse à 0,90 (p<0,001) pour la région Centre-Val de Loire.
Discussion
Cette étude exploratoire a permis de montrer que les données issues de Twitter peuvent être utilisées comme un proxy pour la surveillance de l’épidémie saisonnière de grippe en France. Les résultats indiquent une forte corrélation, à la fois au niveau national et régional, entre les tweets relatifs à la grippe et les consultations pour syndrome grippal rapportées par les réseaux OSCOUR® et SOS Médecins. Ces résultats sont comparables à ceux décrits dans une étude américaine où des corrélations élevées avaient été observées entre les données de Twitter et celles issues des réseaux sentinelles des Centers for Disease Control and Prevention à l’échelle nationale des États-Unis et, dans une moindre mesure, au niveau régional 3.
L’utilisation des médias sociaux comme nouvelles sources de données constitue une avancée pour la surveillance épidémiologique.
Cette approche présente plusieurs avantages : elle ne nécessite pas d’animation de réseau, contrairement aux autres systèmes
décrits dans cette étude. Les données sont gratuites et disponibles en temps réel. Elles apportent un éclairage complémentaire
en fournissant des informations sur un état de santé ressenti par comparaison aux dispositifs existants qui recueillent des
données médicales. Par ailleurs, les mesures d’audience montrent que Twitter est utilisé en majorité par les 16-44 ans (67%) :
ces données permettent donc de décrire le ressenti d’une population de jeunes adultes qui ne sollicitent pas systématiquement
le système de soins. Par rapport à d’autres médias sociaux 15, les données de Twitter offrent également une granularité géographique plus fine, avec un suivi possible des tendances à
l’échelle régionale. Enfin, plusieurs études ont montré que les données issues
de Twitter pouvaient être utilisées à des fins
de prédiction de la dynamique des épidémies de grippe 4,5,6,7,8,9,10,11,12,13,14,15,16.
Cette étude présente néanmoins plusieurs limites. Il n’a pas été possible de savoir si le volume de tweets retourné par notre requête couvrait moins de 1% du volume de tweets publiés et, dans le cas contraire, de mesurer l’impact de l’échantillonnage non-aléatoire de Twitter sur les données analysées. Au final, 22 939 tweets ont été identifiés comme relatifs à l’expression clinique de la grippe sur un volume initial de 198 123. Dans ce contexte, des améliorations doivent être apportées dans le recueil et le prétraitement de ces données. En effet, le recueil des données repose actuellement sur des mots-clés spécifiques (grippe, grippal, grippaux) ; des études récentes ont montré que l’ajout de termes associés à un syndrome grippal (comme par exemples : toux, mal de gorge, maux de tête, etc.) peut améliorer les coefficients de corrélation 17. Si l’implémentation de filtres géographiques a permis de localiser 53% des tweets en France, ce résultat pourrait être encore optimisé en utilisant les informations textuelles contenues dans la description du profil de l’utilisateur ou dans le message même du tweet, ou encore d’inférer la localisation géographique en étudiant les interactions entre les différents utilisateurs (réponse, Retweet, etc.) 18. Dans cette étude, les liens URL et les Retweets ont volontairement été supprimés avant l’application de l’algorithme de classification, conformément à la littérature qui montre que cette étape permet d’augmenter la corrélation des tweets avec les autres sources de données 13. Les résultats du classificateur sur un échantillon aléatoire de 1 000 tweets montrent qu’une grande partie des tweets ont été bien codés (précision de 0,739) ; cependant, 27,5% des tweets codés valides manuellement ont été considérés comme invalides par le classificateur. D’autres techniques d’apprentissage supervisé devront être expérimentées afin d’améliorer les performances du classificateur.
Au cours de cette étude rétrospective, la durée du processus de prétraitement et d’analyse des données de la période d’étude a été évaluée à moins d’une heure. À terme, ce système automatisé pourrait constituer un outil de surveillance des épidémies saisonnières de grippe proche du temps réel, en complément des systèmes existants ou comme alternative à ces derniers s’ils fonctionnaient en mode dégradé (rupture provisoire des flux informatiques). Cependant, l’ensemble du processus nécessite d’être amélioré avant d’être proposé en surveillance de routine. À titre d’exemple, la durée de traitement et d’analyse peut être raccourcie par l’utilisation d’une architecture informatique spécifique (Framework). Parallèlement, une réflexion sur l’utilisation des modèles statiques devra être menée afin d’étudier la possibilité de prédire les tendances futures.
Les courbes épidémiques suggèrent que les données de Twitter pourraient détecter précocement le démarrage de l’épidémie de grippe ; cependant, ce décalage est probablement lié à un biais de sélection. En effet, les données de Twitter (qui est essentiellement utilisé par les jeunes adultes) ont été comparées à celles issues de la population générale et non à la tranche d’âges des 16-44 ans. Une analyse de séries temporelles, en tenant compte de l’âge ainsi que d’autres facteurs comme la circulation d’autres virus responsables d’infections respiratoires hivernales (rhinovirus, virus respiratoire syncytial, etc.), les congés scolaires ou la tendance à long terme devra être réalisée afin d’objectiver ou non ce décalage.
Enfin, il est important de préciser que les tendances observées à partir des données de Twitter doivent être corroborées par des données biologiques. En effet, les médias sociaux sont parfois soumis à des « phénomènes de propagation virale » qui peuvent perturber l’interprétation des données (exemple : effet de panique suite à une information alarmiste non vérifiée).
À notre connaissance, il s’agit de la première étude conduite aux niveaux national et régional afin d’évaluer l’apport des données de Twitter dans la surveillance de l’épidémie saisonnière de grippe en France. Une étude complémentaire portant sur la saison grippale 2017-2018 est prévue afin des confirmer les tendances observées.
Conclusion
L’essor des médias sociaux dans la population française constitue aujourd’hui un véritable challenge pour l’infodémiologie. En effet, les données issues de ces outils de communication peuvent apporter un nouvel éclairage pour la santé publique. Il ne s’agit pas d’opposer cette nouvelle approche aux méthodes traditionnelles mais de les utiliser de manière complémentaire. Cette étude démontre qu’à partir d’un système de recueil, de prétraitement et d’analyse des données de Twitter, il est possible de suivre l’épidémie saisonnière de grippe à l’échelle nationale et régionale tout en complétant les systèmes existants.
Remerciements
Nous remercions Philippe Oesterle et Jean-Bernard Candapanaiken de l’Agence régionale de santé Océan Indien, Céline Caserio-Schöneman de Santé publique France et Luc Vitrant. Remerciements à l’ensemble des partenaires des réseaux OSCOUR® et SOS Médecins.